分析証明書(COA)は、 ページ上部の検索バーにロット番号を入力して検索してください。
安全性データシート(SDS)は、ページ下部の技術資料からダウンロードしてください。
核酸抽出・精製試薬キットのカタログはこちら。
- 日本語クイックマニュアル - AMPure XP プロトコル
- サンプルと磁性ビーズの比率の重要性
- DNA精製試薬キットがシーケンス品質に及ぼす影響
- 自動分注機 Biomek シリーズでの自動化
- Webセミナー:カラム式DNA精製キットとの差異
- お客様の声:iGEM 2021 Kyoto チーム [PDF]
- お客様の声:国立がん研究センター研究所 大木 理恵子 先生 [PDF]
本製品の使用にはマグネットアクセサリが必要です。
製品モデル
OEM パートナーになりませんか? 詳しくはこちら
概要
磁性ビーズ式のAMPure XPは、DNAシーケンス、各種PCR、マイクロアレイ、その他の酵素反応など様々なゲノミック アプリケーションにおけるDNA精製に最適です。SPRI磁性ビーズ技術は、任意の核酸のサイズのみを選択的に磁性ビーズに結合させることが可能です。
![]()
DNA精製用途
![]()
100 bp以上のPCR産物を、高収量で精製
![]()
dNTP、プライマー、プライマーダイマー、塩類、その他夾雑物を効率的に除去
![]()
結合するDNAサイズの制御
精製収量、再現性、スピードを最大化し、NGSワークフロー全体を効率化する AMPure XP は、現代のゲノム アプリケーションの厳しいニーズを満たし、データの損失リスクを最小限に抑えます。イルミナ、オックスフォード・ナノポアテクノロジーズ、PacBio、サーモフィッシャーサイエンティフィックなど主要各社を含め、200を超えるNGSライブラリー調製キットのプロトコルで採用されています。
ワークフローと自動化
AMPure XP ワークフロー
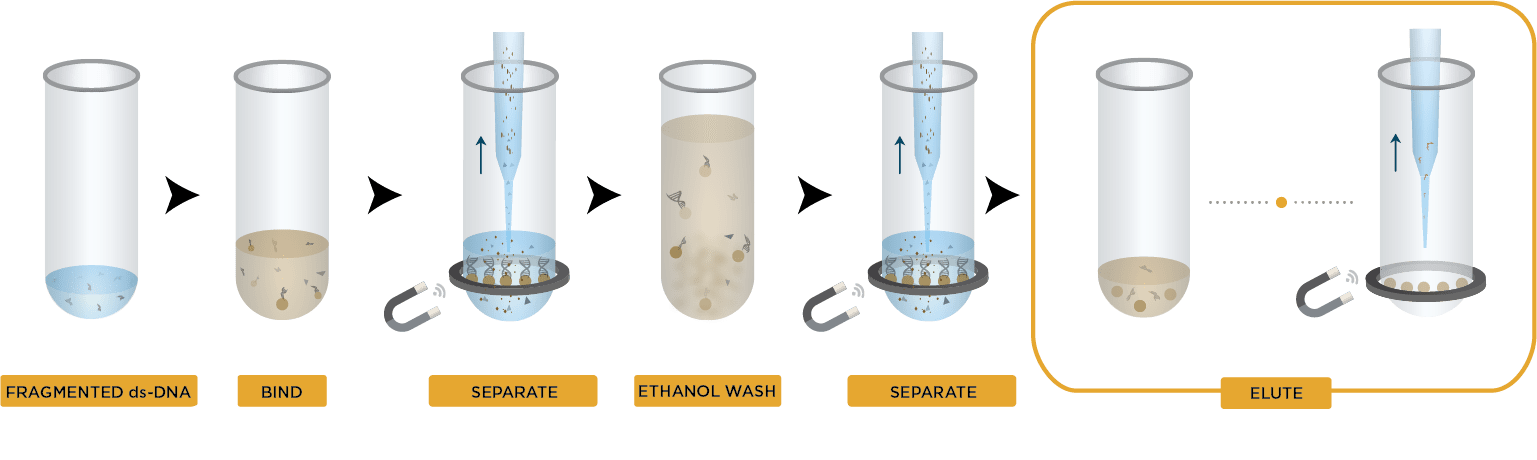
- 磁性ビーズにDNAを結合させる
- 磁石に磁性ビーズを結合させ、夾雑物を除去
- 磁性ビーズを70%エタノールで洗浄し、さらに夾雑物を除去
- 磁性ビーズからDNAを溶出
- 精製DNAを新しいプレートに移す
AMPure XP は、サンプル数に応じて、手動実験または自動分注機を使った自動実験の両方でで行うことができます。
右の表では、48サンプル、96サンプル、384サンプル(96ウェルプレート4枚)の精製を行うのに必要な手作業時間と合計時間を分単位で示しています。
弊社自動分注機 Biomek i5 Multichannel での AMPure XP の自動化の詳細については、アプリケーションノートをご覧ください。
| 手動実験 | 自動実験 | |||
|---|---|---|---|---|
| Batch Size | 48 | 手作業時間 | 25 | 5 |
| 合計時間 | 25 | 22 | ||
| 96 | 手作業時間 | 30 | 5 | |
| 合計時間 | 30 | 22 | ||
| 4 x 96 | 手作業時間 | NR | 10 | |
| 合計時間 | NR | 46 | ||
NR = Not Recommended
仕様
| サンプル | PCR産物、DNA断片 |
| 精製標品 | DNA |
| 収量 | 100 bp以上のPCR産物では通常 60-90%、多くの場合で70-90% |
| ビーズ比率 | 精製にはサンプルの 1.8倍容、その他の比率についてはこちら |
| 様態 | 液体 |
| 容量 | 5 mL, 60 mL, 450 mL |
| アプリケーション | PCR産物精製、DNA精製、NGSライブラリ クリーンアップ |
| 実験形式 | オートメーション、マニュアルともに対応 |
| 基盤技術 | SPRI 磁性ビーズ技術 |
| 保存温度 | 4 °C |
AMPure XP によるDNA精製の自動化方法
手動実験と比較して、Biomek 自動分注機によるAMPure XP 精製プロセスには以下のような利点があります:
- 処理時間短縮とスループットの向上
- セットアップさえすれば精製完了まで完全自動化
- 分注エラーの低減
- 実験の標準化により品質向上
- 実証されたメソッドの迅速な導入
自動分注機 Biomek i5 DNA精製ソリューションについては、メソッドオーバービューをお読みください。
弊社アプリケーションスペシャリストへのご相談は、こちらのフォームから。

よくある質問
他の組織によって開発されたAMPure XPを用いたDNAサイズセレクションのプロトコルも存在しますが、弊社では性能の保証やこのアプリケーションのサポートはいたしません。DNAサイズセレクション専用に開発されたSPRIselectのご使用をお勧めします。AMPure XPとSPRIselectの違いについては、こちらのページをご覧ください。
SPRIselectとAMPure XPの主な違いは、使用目的です。SPRIselectは、ロットごとにDNAセレクションのための厳密なQCを行っているのに対し、AMPure XPはPCR精製やNGSクリーンアップのためのQCを行っています。詳しくはこちらのページをご覧ください。
AMPure XPはDNA/RNAの両方に結合することができますが、RNAClean XPがRNase free条件下で製造・QC確認されているのに対し、AMPure XP はRNase freeを担保していません。
AMPure XPは、ボトルに記載された保存温度で製造・試験されており、その温度でのみ性能を保証することができます。
いいえ。持ち込まれた磁性ビーズは、下流の酵素反応に悪影響を与えません。
可能ですが、溶出量が減ると収量が低下します。また、磁性ビーズが磁石に届かない場合には、磁性ビーズの持ち込みを避けるため、溶出液の一部をウェルに残す必要がある場合があります。磁性ビーズは、必ず溶出液に完全に懸濁してください。
磁性ビーズの核酸結合能は非常に高く、少なくとも核酸 7 μgをAMPure XP 1 μLに結合させることは可能です。しかし、この場合にはDNAにより粘度が高くなり、ピペッティングが困難になります。
収量は、フラグメントサイズ、サンプル量、サンプル濃度、溶出量、その他のいくつかの要因に依存します。理想的な条件下では回収率は90%に達しますが、最適でない条件下では60%にまで低下することもあります。
弊社の磁性ビーズは、結合、洗浄、溶出のための多因子平衡化学に従っています。結合バッファーは、磁性ビーズ表面上の結合部位(官能基)での核酸の結合を促進し、洗浄ステップはこの結合を保ちながら夾雑物を可溶化して除去します。溶出は磁性ビーズと核酸の結合状態を解き、精製核酸が溶液に遊離します。詳細はこちら。
最後のエタノール洗浄を除去した後、溶出バッファーを加える前に、乾燥工程を入れることができます。ゲノムDNAのような高分子量の場合には収量が低下するため、この乾燥工程の実施は推奨されません。40kbまでのDNA断片に関しては、乾燥工程を行うことができます。
ベックマン・コールター ライフサイエンスによる、AMPure XP 技術資料は こちら。
他にもご不明な点ございましたら、お気軽にお問い合わせだください。
引用文献
AMPure XPは、21,000以上の論文で引用(Google Scholar)され、Science、Nature、PNASの論文でも引用されている。ここでは例として2つの論文を紹介します:
Greenwald WW et al. Subtle changes in chromatin loop contact propensity are associated with differential gene regulation and expression. Nat Commun. 2019 Mar 5;10(1):1054. doi: 10.1038/s41467-019-08940-5.
Peffers MJ et al. Transcriptomic profiling of cartilage ageing. Genom Data. 2014 Mar 19;2:27-8. doi: 10.1016/j.gdata.2014.03.001.
技術資料
製品および実証されたアプリケーションは、診断手順での使用を意図したものでも、検証されたものでもありません。