マルチプレックス全ゲノムシーケンシングおよびマイクロバイオームアプリケーションのためのIllumina Nextera XTライブラリ調製の効果的な微量化
Jefferson Lai, John Lesnick, Noël Ruppert
概要
シーケンシングのヌクレオチド当たりの費用は、ここ10 年間で桁違いに下がりましたが、次世代シーケンシングのためのサンプルのライブラリ調製はいまだに費用のかかる工程です。合成生物学において、微生物の全ゲノムシーケンシングは、ゲノム編集の検証、変異誘発のデコンボリューション、ハイスループットDNA 構築の確認、マイクロバイオームサンプルの調査などで標準的なプロセスになりつつあります。本アプリケーションノートでは、IlluminaNextera XT DNA ライブラリキットで使用するリアクションボリュームを微量化し、それによって、標準的なプロトコルで得られるのと同等のデータを維持したうえでリアクションごとの全体的な費用を低減するメソッドの詳細を解説します。次に、この微量化メソッドを様々な種類の微生物でテストした結果、このメソッドが多様なサンプルにおいて効果的であること、また、マイクロバイオーム研究に応用した場合でも、サンプルの多様性、割合、および識別度が維持されることを提示します。
序論
シーケンシングのヌクレオチド当たりの費用は、次世代シーケンシング技術の継続的な向上によって、ここ10 年間で桁違いに下がってきています。しかし、サンプルのライブラリ調製は、いまだにシーケンシングの費用低減のボトルネックとなっています。合成生物学において、全ゲノムシーケンシングは、ゲノム編集の検証、変異誘発のデコンボリューション、ハイスループットDNA 構築の確認、マイクロバイオームサンプルの調査などで標準的なプロセスになりつつあります。Illumina Nextera XT ベースのワークフローを使用して細菌や真菌などの小さいゲノムの配列を決定するための現在の取り組みでは、手作業ではボリュームの微量化が制限されるうえ、処理生成されたごく微量のライブラリのみをシーケンシング分析に使用することになります。Echo 525 リキッドハンドラーをNextera XTキットと組み合わせて使用することにより、過剰なライブラリ生成が低減され、微量でリアクションを得られ、高価な試薬にかかる費用を削減できます。さらに、微量でのタグメンテーションリアクションにより、必要なサンプルgDNA の量も低減されます。本アプリケーションノートでは、タグメンテーションと増幅のリアクションが20分の1 にまで縮小できることを示します。これらのライブラリをIllumina MiSeq ペアエンドシーケンシング分析で マルチプレックスしたところ、同等品質の計量が得られることが示されました。Echo 525 のナノリットルレベルの精度と速度により、Nextera XT ライブラリ調製の精度の高い微量化と、Quant-iT Picogreen 定量分析やAgilentTapeStation 2200 フラグメント分析などのQC 測定が可能になり、その結果、高品質のシーケンシングデータを得ながら、費用を低減し、スループットを向上させることができます。さらに、Echo 525 により、チップを使用するどのリキッドハンドラーとも比べ物にならない速度でシーケンシング用のライブラリをノーマライゼーションしてプーリングすることが可能になります。
メソッドおよびディスカッション
タグメンテーションと増幅のリアクションの微量化
手動分注の微量化は物理的な限界があるため、シーケンシングの質を維持しつつ微量化させることで、サンプル当たりの費用を削減させることを目的として、Echo 525 による分注でのボリュームの条件検討を行いました。この研究には、学術界および産業界で幅広く使用されている大腸菌K-12 ゲノムを選択しました。大腸菌K-12 ゲノムは、代表的な「小さいゲノム」サイズであるため、1 度のMiSeq 分析でライブラリ調製の高度なマルチプレックス条件化が可能です。すべてのサンプルを3 回繰り返し分析しました。表1に示す通り、異なるボリュームのgDNA およびタグメンテーション試薬を、25 μL 以外はすべてEcho 525 で分注しました。さらに、各条件のリアクションに合わせたボリュームのTD+ATM バッファーもEcho 525 を用いて分注しました(25 μL のみ手動分注)。55 ℃のタグメンテーションインキュベーションはThermoFisher ProFlex サーマルサイクラーを用いて行いました。リアクションボリュームによって加熱に必要な時間が異なるため、液量ごとにPCR プレートを分けて実験を行いました。タグメンテーションを中和するためのNT バッファーはEcho 525 で分注し、タグメンテーション後のライブラリ、インデックス付きプライマー、Nextera PCR Mix (NPM) もEcho 525 を用いて各リアクションボリュームに適した量を分注し、Nextera XT PCR の手順に従って増幅を実行しました。次に、各ライブラリをPicogreen アッセイで定量化しました。このアッセイでは、Echo 525 を使用してサンプルおよびPicogreen 試薬を微量化した20 μL のボリュームでGreiner 384 ウェルプレートに分注し、BMG PHERAstar 上で結果を読み取りました。各ライブラリに、TapeStation 2200 を通じてフラグメントサイズ分析も行いました。Echo 525 を使用して、サンプルと試薬をアッセイプレートに分注しました。定量化データと平均フラグメントサイズデータを組み合わせて使用して、ノーマライゼーションワークリストを生成しました。Echo 525 を用いて微量のライブラリを正確かつ精密に1 つのウエルに移すことにより、複数のライブラリのノーマライゼーションとプーリングをワンステップで行えます。プーリング後、ライブラリを水酸化ナトリウムで変性させ、Illumina MiSeq でペアエンド300 リードシーケンシングを行いました。その後、データをBiomatters Geneious ソフトウエアで解析し、ゲノムアライナーを使用してNCBI から大腸菌K-12 MG1655 基準ゲノムにアラインメントしました。結果のまとめを以下の表に示します。
| インプット DNA | タグメンテーション ボリューム |
PCR ボリューム | フラグメント サイズ |
カバレッジ | 平均Qスコア | > Q30 | |
| 平均 | StDev | ||||||
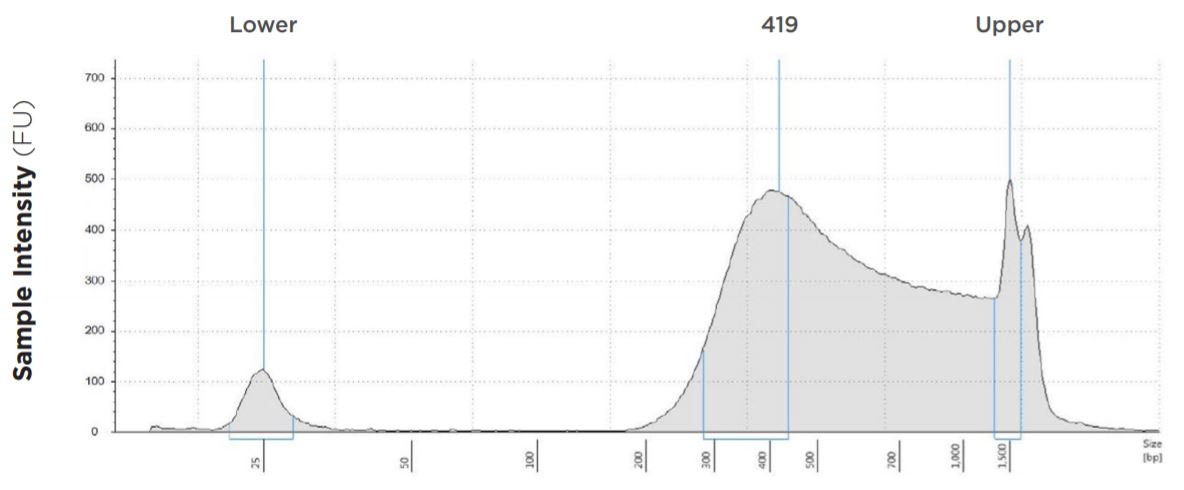
| 1 ng | 25 µL | 25 µL | 419 bp | 9 | 4.5 | 33.7 | 82.70% |
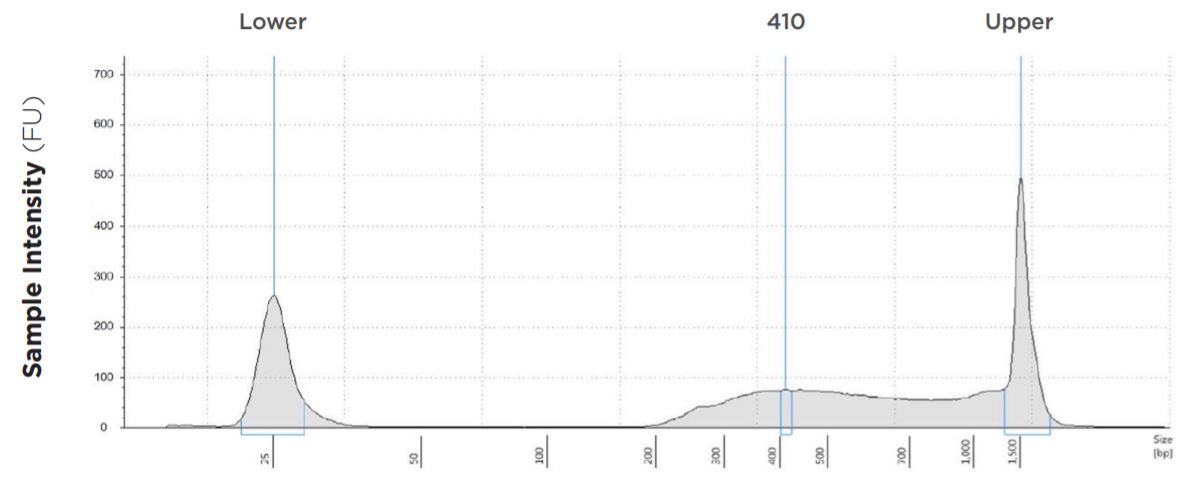
| 1 ng | 25 µL | 25 µL | 410 bp | 8.5 | 4.6 | 33.5 | 81.80% |
| 0.5 ng | 12.5 µL | 6.25 µL | 372 bp | 8.4 | 4.6 | 34.4 | 85.50% |
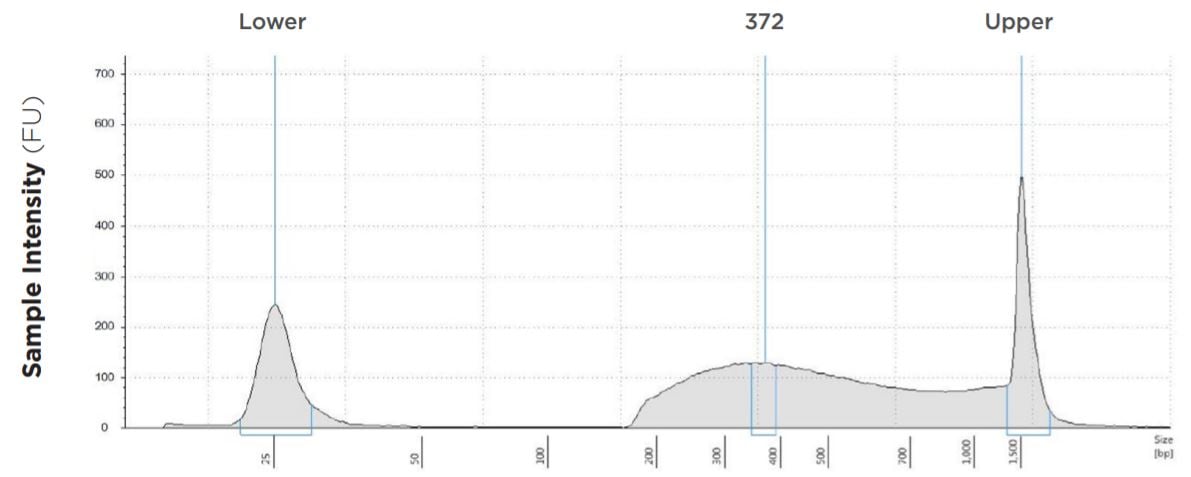
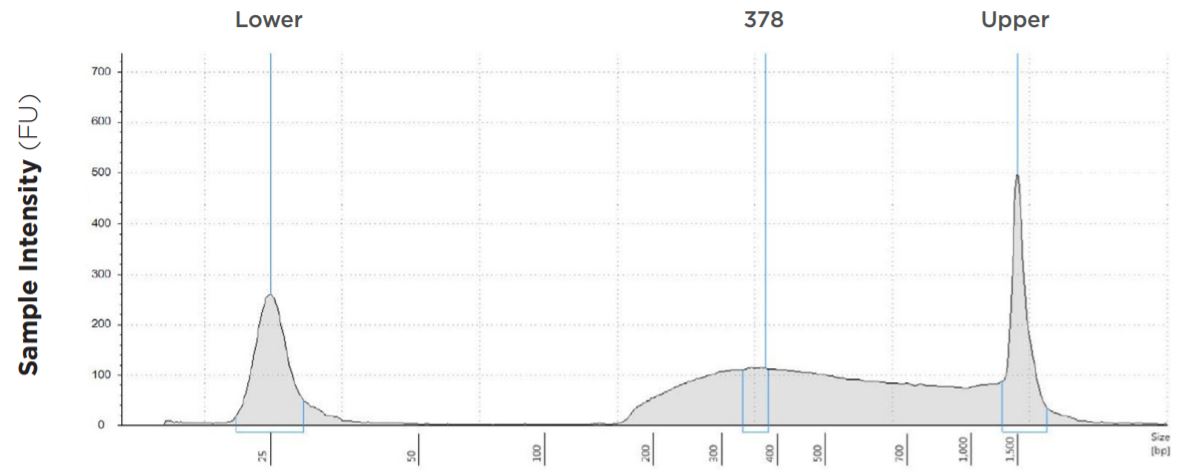
| 0.25 ng | 6.25 µL | 6.25 µL | 378 bp | 9.4 | 5 | 33.9 | 83.70% |
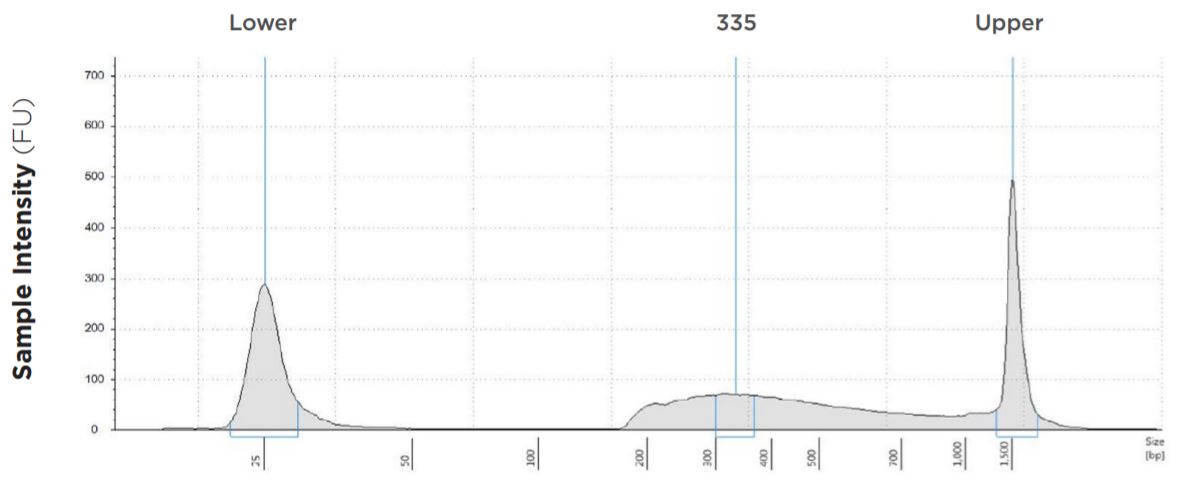
| 0.05 ng | 1.25 µL | 2.5 µL | 335 bp | 9.7 | 5.5 | 34.1 | 84.40% |
表1:個々のライブラリ条件および得られた結果一覧。
25 μL タグメンテーション / 25 μL PCR
図1A:表1- ①のTapeStation 電気泳動図
25 μL タグメンテーション / 2.5 μL PCR
図1B:表1- ②のTapeStation 電気泳動図
12.5 μL タグメンテーション / 6.25 μL PCR
図1C:表1- ③のTapeStation 電気泳動図
6.25 μL タグメンテーション / 6.25 μL PCR
図1D:表1- ④のTapeStation 電気泳動図
1.25 μL タグメンテーション / 2.5 μL PCR
図1E:表1- ⑤のTapeStation 電気泳動図

図2:Picogreen 定量化の結果。20 pg/μL ~1 μL のラムダDNA を使用し検量線を作成した。定量の結果は、TapeStation フラグメント解析と合致した。収量減少は、PCR 容量の減少が影響していると思われる。 すべての条件において再現性が高く、Tag25/PCR25 を手動(マニュアル)で行った場合、より大きな変動が見られた。
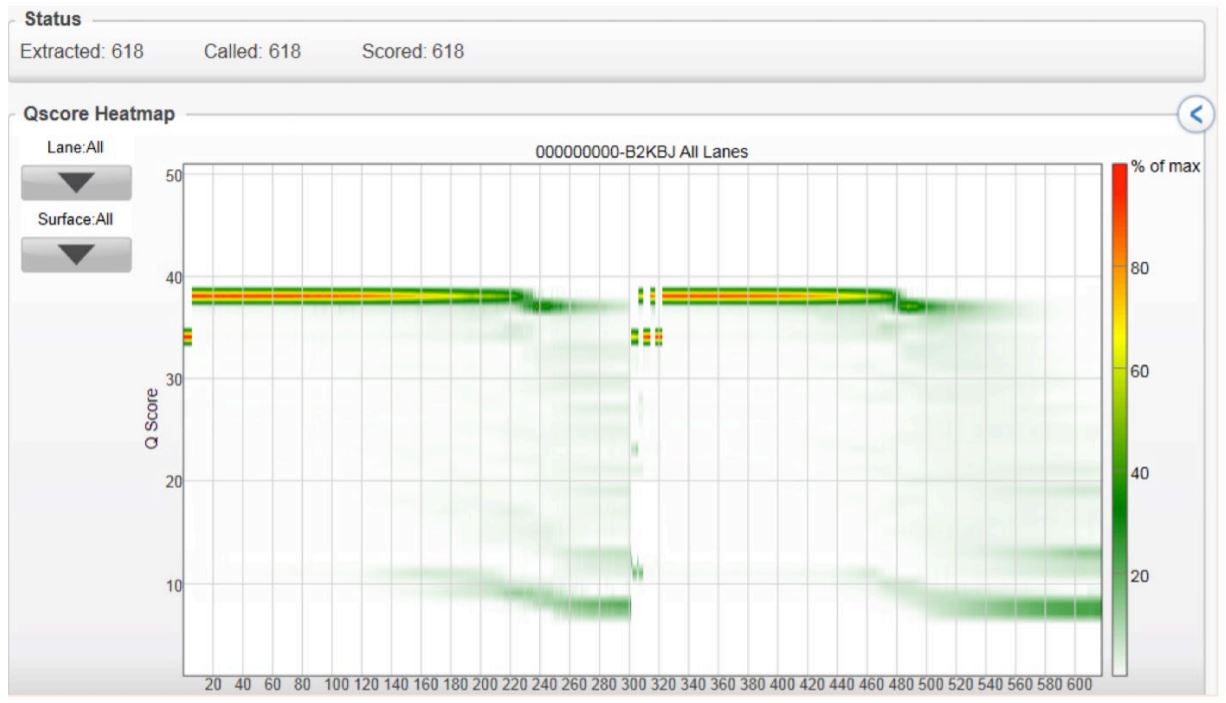
図3: MiSeq を用いたマルチプレックス解析のQ スコア。さまざまなリアクションボリュームにおいて従来の2x300 ゲノム分析と同等のQ スコアが得られた。
RefSeq に対するアラインメントカバレッジ
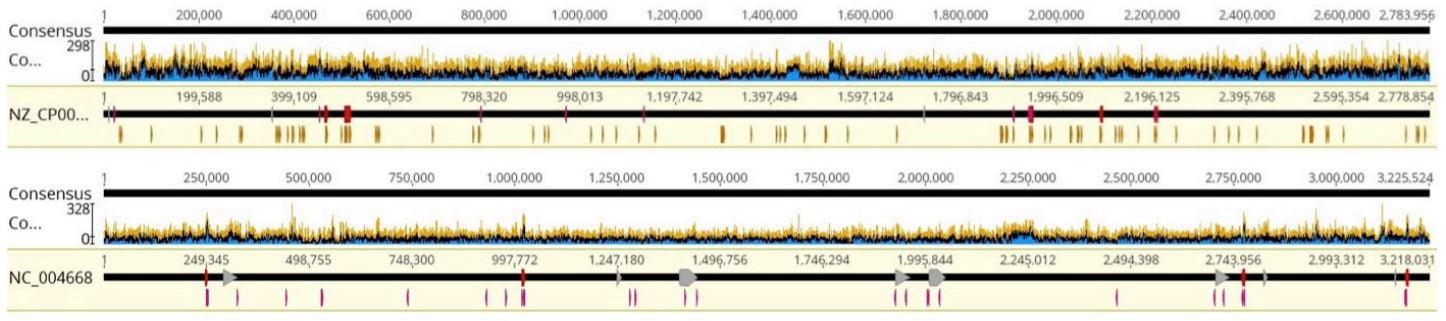
図4: Geneious のアラインメントグラフ。カバレッジは、使用したリアクションボリュームに関わらず均一。大腸菌K-12 gDNA(ATCC)はNCBI 大腸菌K-12 MG1655 基準ゲノムにアラインメント。
実験の過程で、微量化したリアクションボリュームでもフラグメンテーションは有効であることを発見しました。小さいボリューム増幅リアクションとそれに続くPCR クリーンアップで確実に少ないライブラリが生成されますが、Echo 525 のPicogreen の定量化データに基づいて短時間で標準化することにより懸念点は払拭できます。ライブラリのQC 中に、すべてのボリュームのタグメンテーションでよく似たフラグメントの平均長とサイズの分散が得られることを発見しました。シーケンシングの後、手動ワークフローをEcho 525 の微量化ワークフローと比較したところ、カバレッジ、標準偏差、およびQ スコアに大きな影響はないことが認められました。これにより、データを劣化させることなくリアクションボリュームを減らすことが可能になり、その結果、試薬にかかる費用とインプットDNA を節約することができます。この実験により、続く生産実験のために2.5 μL/5 μL のタグメンテーション/PCR リアクションボリュームを使用することの確実性が得られました。これらのボリュームは、当ラボの設備と機器での対応しやすさから選択したものです。
高スループットのライブラリ調製と全ゲノムシーケンシングのマルチプレックス
学術的/ 産業的な稼働状況を踏まえ、一般によく研究され、多様性を持つ9 種類の微生物とMiSeq 上で一緒に分析するための1 種類のマイクロバイオームサンプルを選びました。ゲノムサイズとMiSeq v3 試薬キットで得られる出力に基づいて、これらのサンプルをマルチプレックス法を用いて、統計的信頼度の高いカバレッジを得ることができました。我々は研究開始当初から、微量化したワークフローの堅牢性をさまざまなゲノムサイズ、GC 含量、およびゲノムの組織化でテストすることを目的としていました。また、マイクロバイオームサンプルはこのアプリケーションで一般によくテストされている10 種類の微生物を含み、この規模での微生物と菌株の同定が可能かどうかを突き止め、微生物の度数がプロセス全体で終始維持されるかどうかを突き止めることを模索しました。各微生物のサンプルgDNA をまず手動で濃度5 ng/μL へと標準化しました。次に、前述の実験で説明したメソッドで、今度は2.5 μL/5 μL タグメンテーション/PCR リアクションボリュームを使用して、ライブラリ調製手順を実施しました。その後、SPRI ビーズクリーンアップを使用し、0.6x の比率で、ライブラリを手動でクリーンアップし、Picogreenアッセイを通じて定量化し、TapeStation 2200 で解析しました。定量化データと平均フラグメントサイズデータを組み合わせて使用して、ノーマライゼーションワークリストを生成しました。Echo 525 を用いて、微量のライブラリを正確に1 つのウェルに移すことにより、複数のライブラリを同時に標準化してプールすることができます。このプールしたライブラリを、その後、Qubit を通じて定量化し、4nM に標準化し、水酸化ナトリウムで変性させてから、ペアエンド300 bp リードのためにIllumina MiSeq にロードしました。各サンプルを対応するNCBI 基準ゲノムまたは近いゲノムにアラインメントし、Biomatters Geneious ソフトウエアとGeneious アライナーを使用してシーケンシングデータを計算しました。
| 種 | ゲノムサイズ | MiSeq インデックス比率 |
RefSeqへの アラインメント |
カバレッジ | StDev | 信頼スコア | フラグメント サイズ |
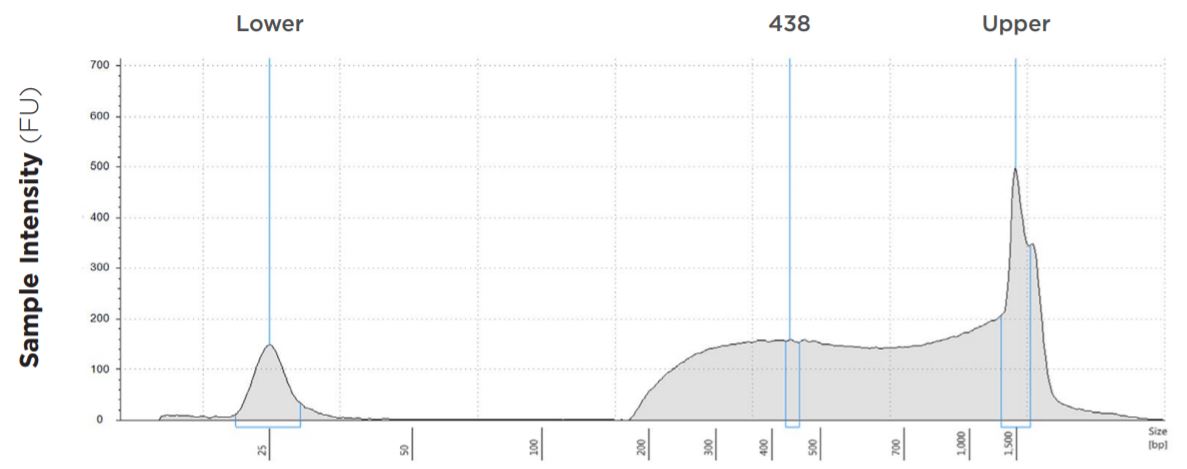
| サルモネラ菌 | 4.70 Mbp | 5.94% | 98.3% | 53.8x | 23.6 | 34.7 | 438 bp |
| 大腸菌 | 4.65 Mbp | 1.20% | 99.40% | 12.5x | 6 | 33.2 | 504 bp |
| 緑膿菌 | 6.34 Mbp | 1.01% | 93.80% | 7.9x | 4.8 | 31.1 | 481 bp |
| サッカロマイセスセレヴィシエ | 12.16 Mbp | 9.36% | 99.80% | 34.3x | 21.9 | 34.7 | 420 bp |
| ピキアパストリス | 9.59 Mbp | 1.59% | 99.60% | 9.9x | 7.5 | 34.2 | 436 bp |
| クリプトコッカスネオフォルマンス | 18.91 Mbp | 3.45% | 98.40% | 10.4x | 5.6 | 33.7 | 512 bp |
| リステリアモノサイトゲネス | 2.95 Mbp | 5.51% | 91.60% | 80.1x | 47.7 | 35.3 | 401 bp |
| 黄色ブドウ球菌 | 2.8 Mbp | 4.30% | 99.99% | 83.3x | 40.3 | 35.3 | 444 bp |
| エンテロコッカスフェカリス | 3.36 Mbp | 3.55% | 99.94% | 61.1x | 31.1 | 34.8 | 457 bp |
| マイクロバイオーム | 6.07% | 396 bp |
表2:マルチプレックス2x300 MiSeq 分析で含まれていた各サンプルの要約データ。
サルモネラ菌
図5:サルモネラ菌(Salmonella enterica)のライブラリ準備および磁気ビーズクリーンアップが終わった後のTapeStation 電気泳動図。代表サンプル。0.6x SPRI ビーズを使用してサイズ選択カットオフを実施。フラグメントの平均長は約438 bp、右側テーリングあり、2x300 リード。

図6:アッセイのセットアップにEcho 525 を使用してライブラリ調製とSPRI ビーズクリーンアップを終えた後のサンプルのPicogreen 定量化データ。サンプルごとの収量は変わる一方で、Echo 525 を用いることで標準化でき、1 分間以内にサンプルを一緒にプールすることができた。
図7:a)黄色ブドウ球菌および b)エンテロコッカスフェカリス、代表サンプルのカバレッジグラフ。
今回のサンプル中3 サンプルでは、シーケンシングした特定の菌株/ 血清型はNCBI 基準シーケンスにはありませんでした。これらのケースでは、近縁の種を使用しました。これより、マッピングにおける低い方の値が基準になることがわかります(RefSeq へのアラインメント)。この3 種類のサンプルは、サルモネラ菌(Salmonella enterica)、緑膿菌、リステリアモノサイトゲネスです。それ以外は、ほぼすべてのリードがその対応する基準にマッピングされ、ゲノムは良好に網羅されており、信頼スコアも高く示されました。さまざまなゲノムサイズ、GC 含有量、およびゲノムの組織化の全体にわたって同様の結果が得られました。カバレッジは、サンプルに割り当てられているリードの数(MiSeq Index Representation)とカバーする必要のあるベースペアの数(ゲノムサイズ)両方の関数であり、カバレッジのサンプル間での変動は当初から予測されたものでした。さらなる最適化と標準化を行うことで、サンプル全体にわたるカバレッジをさらに統一することが可能だと考えられます。全体的な結果として、Echo 525 の微量化ワークフローによって、試薬にかかる費用とインプットを減らしながらも、さまざまな微生物全体にわたって高い品質の全ゲノムシーケンシングデータを生成できることが示されました。
マイクロバイオーム
前述の実験でのマイクロバイオームサンプルの目的は、Echo 525 でのライブラリ調製ワークフローで、微生物を同定する機能が保持されるかどうか、そして最初に供給されたときの割合を維持することができるかどうかを見極めることでした。これをテストするために、Zymo Research から微生物群のDNA 標準を入手し、それを前述の実験中にその他のサンプルと一緒に処理しました。マイクロバイオームサンプルには、一対のインデックスを使用してバーコードを付けました。デマルチプレックスの後、リードの合計数をこのサンプルから取得し、Biomatters によるGeneious ソフトウエアでGeneious アライナーを使用して、リードをマイクロバイオーム内に含めようとしている各微生物にアラインメントしました。成功したアラインメントを全体の割合として計算し、その後、既知のgDNAの存在量と比較しました。
| ソース | 微生物 | レファレンス GC |
測定GC | gDNA (%) | リード | 実測分布 (%) |
| ZymoBiomics D6305 | 緑膿菌 | 66 | 67 | 12 | 209,673 | 14 |
| 大腸菌 | 57 | 53 | 12 | 242,562 | 16 | |
| サルモネラ菌 | 52 | 53 | 12 | 297,457 | 20 | |
| 乳酸菌 | 53 | 53 | 12 | 162,806 | 11 | |
| エンテロコッカスフェカリス | 38 | 41 | 12 | 113,919 | 8 | |
| 黄色ブドウ球菌 | 33 | 38 | 12 | 126,185 | 8 | |
| リステリアモノサイトゲネス | 38 | 41 | 12 | 124,416 | 8 | |
| 枯草菌 | 44 | 46 | 12 | 179,820 | 12 | |
| サッカロマイセスセレヴィシエ | 38 | 39 | 2 | 27,137 | 2 | |
| クリプトコッカスネオフォルマンス | 48 | 48 | 2 | 46,946 | 3 | |
| 1,487,546 | ||||||
表3:その既知の値に対して正常にアラインメントできたリード。
マイクロバイオームサンプル
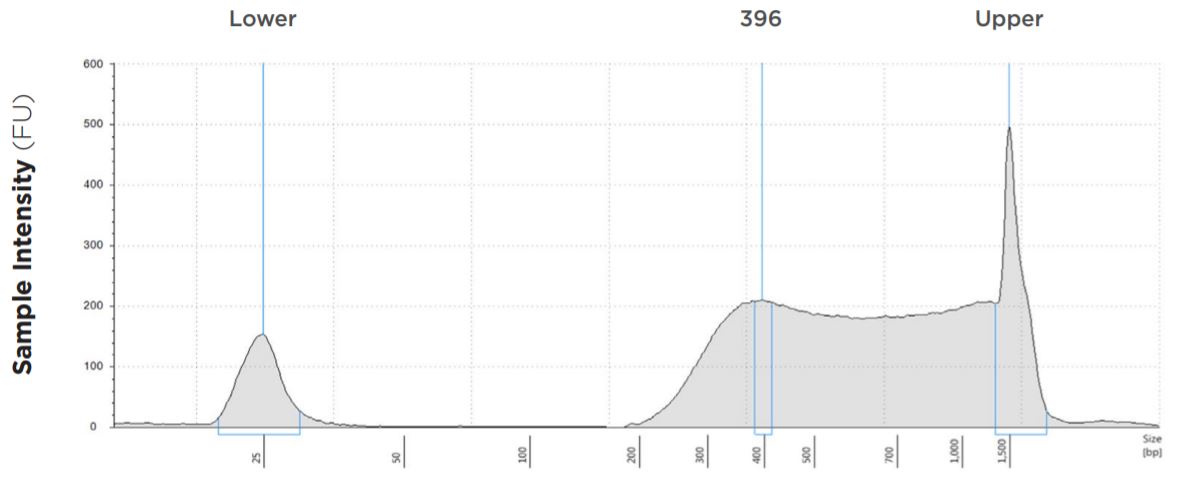
図8:ライブラリ調製および磁気ビーズクリーンアップ後のTapeStation 電気泳動図。0.6x SPRI ビーズを使用してサイズ選択カットオフを実施。フラグメントの平均長は約396 bp、右側テーリングあり、2x300 リード。
微量化したライブラリ調製とマイクロバイオームサンプルのシーケンシングの後、リードは、その対応する微生物と実測分布を反映する割合でマッピングされることがわかりました。マイクロバイオームの同定と個体群調査の目的においては、通常はGC が偏る酵素リアクションにも関わらず、微量化したライブラリ調製によって有意な偏りは導入されません。このメソッドは、微生物および注目する遺伝子の多様性と個体群を探るためのマイクロバイオームの研究およびメタゲノム調査に使用できます。
結論
ここでは、Illumina Nextera XT DNA ライブラリ調製キットのプロトコルで使用するリアクションボリュームを微量化するメソッドについて詳しく説明し、これを小さいさまざまなゲノムサンプルならびにマイクロバイオームサンプルに適用しました。また、Quant-iT Picogreen アッセイ、Agilent TapeStation 2200 フラグメント解析などの関連するQC 計測のプロトコルについても説明しました。最後に、ライブラリの標準化とプーリングにEcho 525を使用することによる影響について取り上げました。
分注ボリュームを25 nL にまで減らすと、試薬ボリュームを明らかに減らすことができ、その結果、研究にかかる費用が低減し、同等の試薬量でよりスループットを向上できます。今回の場合、手動プロトコルのリアクションボリュームと比べた場合に、カバレッジ、標準偏差、および信頼スコアに有意な影響を発生させることなく、タグメンテーションとPCD リアクションのためのボリュームが20 分の1 に低減できました。
費用の低減に加えて、Echo シリーズのリキッドハンドラーの柔軟性とウェル間で分注できる機能により、アッセイの最適化も容易に行えるため、単一のソースプレートから幅広いさまざまな条件をテストできます。タグメンテーションと増幅プロトコルの微量化により、定められた範囲のボリュームで一連の実験を簡単に実施することができ、最終的に最適化された2.5 μL/5 μL タグメンテーション/PCR リアクションボリュームを特定するに至りました。
25 nL 刻みでの極めて正確な微量分注により、サンプルの標準化とプーリングを単一ステップで行うことが可能になり、その結果、チップを使用して行う標準的な分注では達成不可能だった時間の節約が可能になります。
Echo 525 は、幅広いさまざまな微生物にわたるライブラリ調製の微量化を増進し、変化し続ける系統、GC 含有量、ゲノムサイズ、ゲノム組織においても、全ゲノムシーケンシングの一貫した高品質な結果を提供します。微量化されたプロセスではサンプルの多様性、割合、および識別度が維持されるため、このメソッドは、マイクロバイオームおよびメタゲノムアプリケーションにも応用できます。
| 機器 | 製造元 |
| Echo 525リキッドハンドラー | ベックマン・コールター ライフサイエンス |
| Allegra X-14 遠心分離機 | ベックマン・コールター ライフサイエンス |
| MixMate | Eppendorf |
| Qubit | ThermoFisher |
| TapeStation 2200 | Agilent |
| BMG PHERAstar | BMG Labtech |
| ProFlex PCR System | ThermoFisher |
| 384 ウェルポストマグネットプレート | Alpaqua |
| MiSeq シーケンシング装置 | Illumina |
| 試薬 | 製造元 | 部品番号 |
| Nextera XT DNA 96-Sample Prep Kit | Illumina | FC-131-1096 |
| Nextera XT Index Kit v2 Set A | Illumina | FC-131-2001 |
| PhiX Control v3 | Illumina | FC-110-3001 |
| TapeStation D1000 HS キット | Agilent | #5067-5584, #5067-5585 |
| Qubit dsDNA HS アッセイキット | ThermoFisher | #Q32851 |
| Quant-IT Picogreen dsDNA アッセイキット | ThermoFisher | #P11496 |
| Agencourt AMPure ビーズ | ベックマン・コールター ライフサイエンス | #A63881 |
| 200 プルーフエタノール | Sigma Aldrich | #E7023 |
| MiSeq 試薬 Kit v3 (600 サイクル) | Illumina | MS-102-3003 |
| サルモネラ菌 | ATCC | BAA-1740D-5 |
| 大腸菌 | ATCC | 10798D-5 |
| 緑膿菌 | ATCC | 9027D-5 |
| サッカロマイセスセレヴィシエ | ATCC | 204508D-5 |
| ピキアパストリス | ATCC | 28485D-5 |
| 枯草菌 | ATCC | 82D-5 |
| クリプトコッカスネオフォルマンス | ATCC | 208821D-2 |
| リステリアモノサイトゲネス | ATCC | 19114D-5 |
| 黄色ブドウ球菌 | ATCC | 25923D-5 |
| エンテロコッカスフェカリス | ATCC | 700802D-5 |
| ZymoBiomics マイクロバイオーム | Zymo Research | D6305 |
| 消耗品 | 製造元 | 部品番号 |
| 384 ウェルPP マイクロプレート | ベックマン・コールター ライフサイエンス | 001-14555 |
| 384 ウェルLDV プラスマイクロプレート | ベックマン・コールター ライフサイエンス | 001-12782 |
| TapeStation プレート | Agilent | #5067-5150 |
| Qubit マイクロチューブ | ThermoFisher | #Q32856 |
| 384 ウェルPCR プレート | Bio-Rad | #HSP3805 |
| 384 ウェルブラックフラットクリアボトムマイクロプレート | Greiner | #781096 |
| 1.5 mL DNA LoBind チューブ | Eppendorf | #022431021 |