20カラー イムノタイピングデータのマシンラーニング アルゴリズムを用いた細胞サブセットの詳細解析
目的
本アプリケーションノートではCytoFLEX LXによる20カラーパネルのマシンラーニングを用いた解析について紹介します。データの可視化にはクラウドプラットフォームサイトメトリー解析ソフトウエアCytobankのviSNE・FlowSOM・SPADEを用いました。従来のマニュアルゲーティングにはサイトメトリー解析ソフトウエアKaluzaを用い、CytobankのFlowSOMによる教師なしクラスタリング結果の細胞サブセットと比較しました1,2。マシンラーニングで多く採用されている教師なしアルゴリズムは、従来の既知集団のマニュアルゲーティングよりもバイアスを排除したサブセットの同定や、新規サブセットやゲーティングロスによって見落とされていたサブセットの発見を可能とします。ハイパラメータ(高次元または多色)データセットの詳細な解析にはこれまでの単純なデータセット解析に比べ多くの時間を必要としますが、マニュアルゲーティングと比較してマシンラーニングでは操作時間を短縮できる点においても優れています。
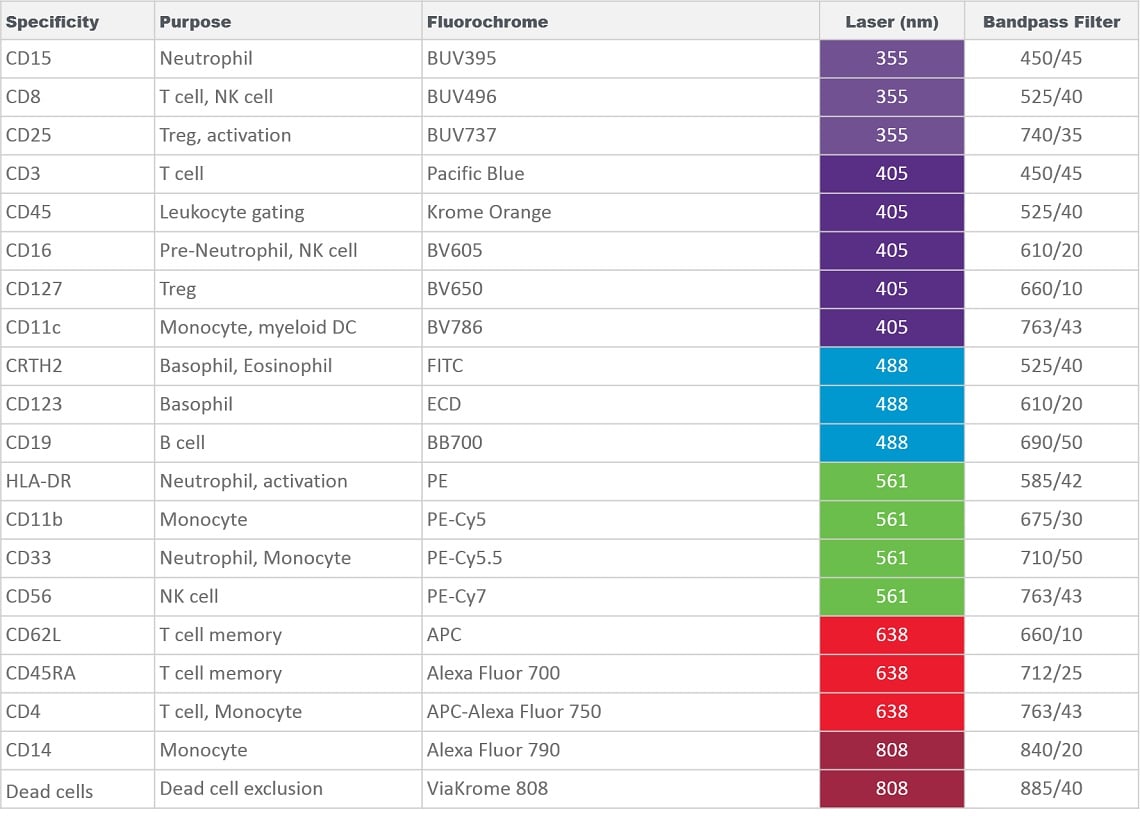
血液サンプルをTable 1 に示す20カラーパネル抗体で染色した後、Versalyse(PN: IM3648)のスタンダードな手順で赤血球を溶血し、染色したサンプルをCytoFLEX LX 6レーザー (PN: C40324)で測定しました。フィルタ構成は、用いた蛍光色素の同定に最適になるよう設定しました。
Table 1. 20カラーパネル
コンペンセーションとlogicleスケールへの変更をKaluzaで行い、コンペンセーション・スケーリング済データとしてCytobankへエクスポートしました。エクスポートにはKaluza Cytobank Pluginを使用しました。Kaluzaはマニュアルゲーティングによるポピュレーション解析にも用いられました。
マシンラーニングによるデータ解析の準備として、デブリス(細胞以外のゴミ)やダブレット、死細胞などの解析対象外のイベントを除きます。これらのイベントは結果のプロットに良くない影響を与え、また統計結果に混乱を招きかねないため、適切に除外される必要があります。研究や解析対象によっては、プレゲーティングによって対象とする細胞集団を絞った方が良い場合もあります( Figure 1)。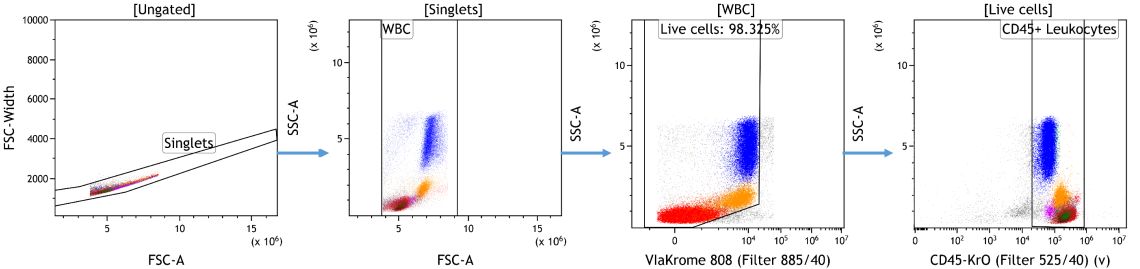
Figure 1. Kaluzaによるデータのクリーンアップ。ダブレット除去をFSC-A vs FSC-widthで行なった後、FCS vs SSCで白血球(WBC)にゲーティングした。ViaKrome 808 陰性を生細胞として同定し、CD45陽性をLeukocyteとした。プロットはイメージ目的。
Kaluzaを用いて、ヒト末梢血の細胞サブセット同定を既知の知見に基づいて3 マニュアルゲーティングしました。
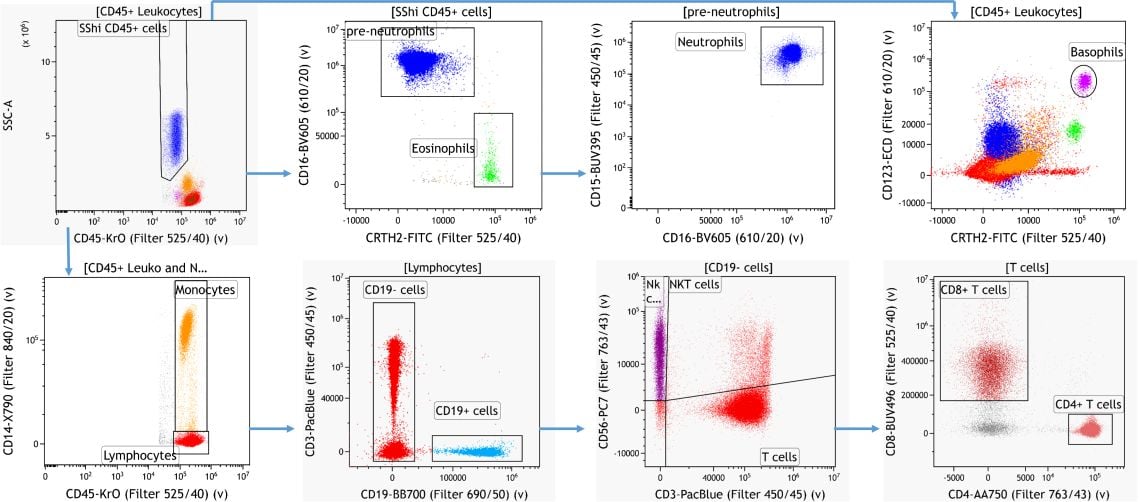 Figure 2. 既知情報に基づくLeukocyteサブセットの同定。Kaluza を使用し、プロットはイメージ目的。
Figure 2. 既知情報に基づくLeukocyteサブセットの同定。Kaluza を使用し、プロットはイメージ目的。
パラメータまたは蛍光チャネル(色)を次元として捉え、マシンラーニングツールを用いた場合、バイアスや知見なしでも細胞サブセットの同定を行うことが可能となります。高次元データ内の情報をviSNEのような次元圧縮アルゴリズムによって2次元プロットに可視化することができるようになります1。FlowSOM等のクラスタリングアルゴリズムはマーカー発現の類似する細胞を自動的クラスターというグループに同定することを可能とします2。
本アプリケーションノートのデータセットでの更なる解析のため、Figure 2のマニュアルゲーティングに用いたマーカーにてviSNEによる次元圧縮を行いました。マニュアルゲーティングに用いたマーカー11 種類(CD45, CRTH2, CD123, CD15, CD14, CD16, CD56, CD3, CD4, CD8, CD19) の情報、つまり11 次元の情報は、viSNEの次元圧縮によって2 次元マップ上に可視化されています。ヒトの認知機能で直感的に把握しやすい2次元に高次元情報を落とし込むことで、迅速な判断と複雑な結果をディスカッションをするためのグラフィック化(可視化)をすることができるようになります。サイトメトリーデータでは特に、データの各細胞/イベントの生物学的な分類に役立ちます。フェノタイプの類似した細胞/イベントはお互いに近い位置にプロットされ島状の分布を展開します。結果のviSNEマップの等高線プロットをFigure 3Aに示します。
次元圧縮の後、データ内の細胞/イベントを12 個のメタクラスターと呼ばれるグループに自動的にクラスタリングするため、FlowSOMを実行しました。細胞集団の同定に用いるマーカーでFlowSOMを行い、その結果をviSNEマップ上にオーバーレイ表示すると、解析クオリティの確認を容易にすることができます。もし最適な結果を得るためにアルゴリズム設定のさらなる調整が必要となったときには、どのような調整が必要かを検討するための基準やヒントになります。
Figure 3Bでは、FlowSOMによるメタクラスターをviSNEマップ上にオーバーレイ表示しています。島状の分布とメタクラスター分布(オーバーレイカラーの違い)が良く一致していることがわかります。各メタクラスターのフェノタイプを同定するには、Figure 3Cのようなヒートマップ表示を用いるととても簡単になります。
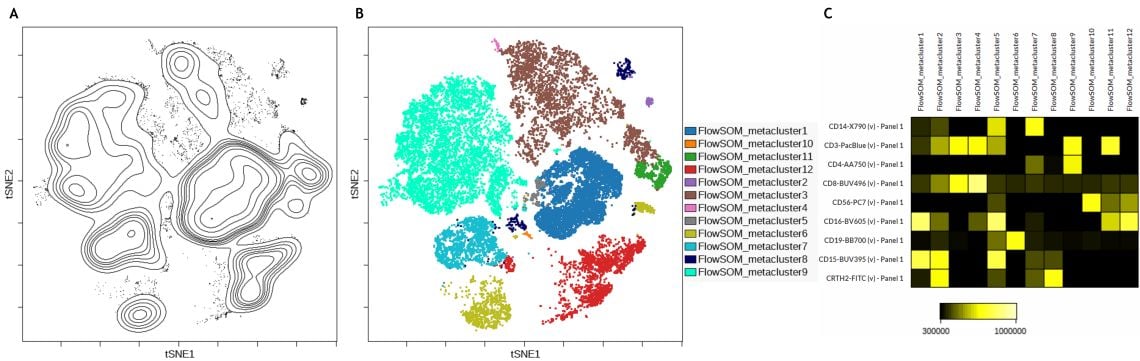 Figure 3. viSNE、FlowSOM、ヒートマップを用いた細胞サブセットの同定。A)viSNEマップの等高線プロット B)viSNEマップ上に展開したFlowSOMメタクラスターのオーバーレイプロット C)各FlowSOMメタクラスターのマーカー発現強度ヒートマップ表示。データはKaluzaによってコンペンセーションとlogicleスケーリング済で、Kaluza Cytobank PluginによってCytobankクラウドプラットフォームへアップロードされた。viSNE設定はマニュアルゲーティングで細胞集団を同定するのに用いられた11 マーカー、iterations = 3,000、perplexity = 30、theta =0.5。FlowSOM設定は以下のとおり。Clusering channel = viSNEと同じ、Clustering method = Hierarchical Consensus、Number metaclusters=12、Number clusters = 121。プロットはイメージ目的。
Figure 3. viSNE、FlowSOM、ヒートマップを用いた細胞サブセットの同定。A)viSNEマップの等高線プロット B)viSNEマップ上に展開したFlowSOMメタクラスターのオーバーレイプロット C)各FlowSOMメタクラスターのマーカー発現強度ヒートマップ表示。データはKaluzaによってコンペンセーションとlogicleスケーリング済で、Kaluza Cytobank PluginによってCytobankクラウドプラットフォームへアップロードされた。viSNE設定はマニュアルゲーティングで細胞集団を同定するのに用いられた11 マーカー、iterations = 3,000、perplexity = 30、theta =0.5。FlowSOM設定は以下のとおり。Clusering channel = viSNEと同じ、Clustering method = Hierarchical Consensus、Number metaclusters=12、Number clusters = 121。プロットはイメージ目的。
各細胞/イベントのチャネルの強度をカラーで表示するviSNEマップドットプロットは、viSNEマップ上に分布する各島が、どのようなマーカー発現パターンをもとに形成されているのかを理解するのに役立ちます。Figure 4では、同一のviSNEマップ上でカラー表示するチャネルをCD19、CD4、CD8に切り替えたものを並べて表示するとともにFlowSOMメタクラスターのオーバーレイと比較します。
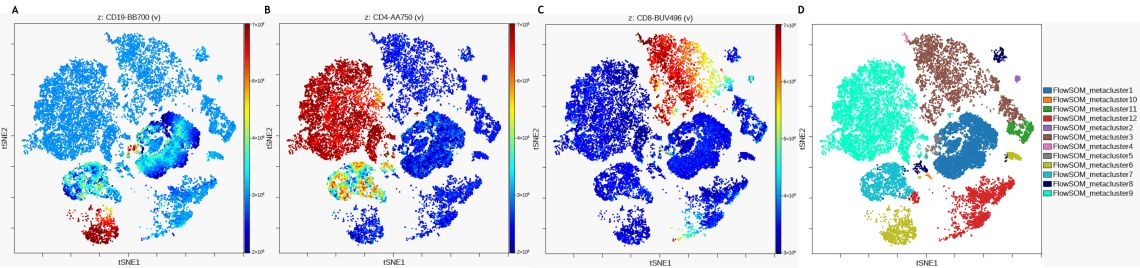
Figure 4. 同一viSNEマップでの細胞集団同定マーカー発現レベルのカラー表示。A)CD19 B)CD4 C)CD8 D)viSNEマップでのFlowSOMメタクラスターオーバーレイ表示。データはKaluzaでコンペンセーションとlogicleスケーリング( リニアとLogのハイブリッドスケール)を済ませた後、Kaluza Cytobank PluginでCytobankへアップロードした。viSNE設定はマニュアルゲーティングで細胞集団を同定するのに用いられた11マーカー、iterations = 3000、perplexity = 30、theta =0.5。FlowSOM設定は以下のとおり。Clusering channel = viSNEと同じ、Clustering method = Hierarchical Consensus、Number metaclusters=12、Number clusters = 121。プロットはイメージ目的。
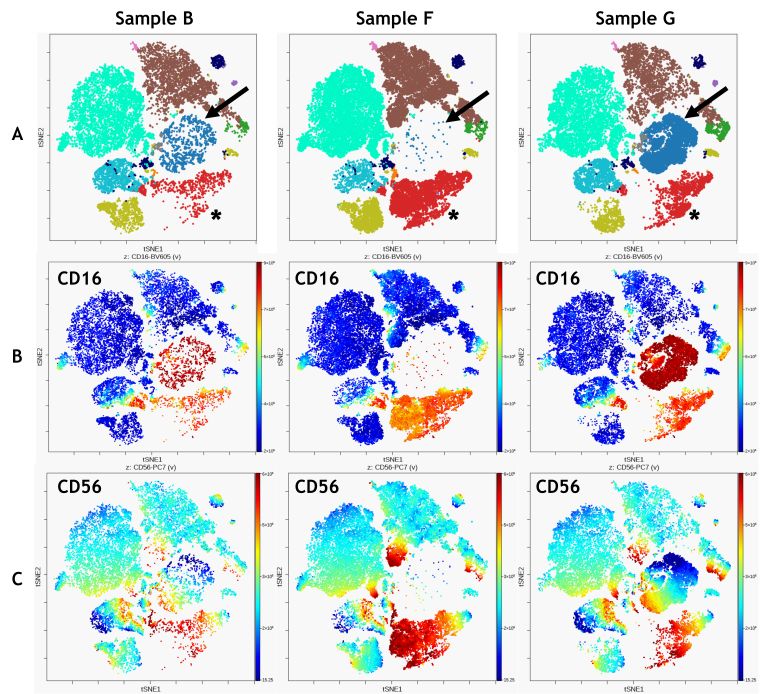
Figure 5. 3つのサンプルの比較。A)viSNEマップ上へのFlowSOMメタクラスターオーバーレイ表示。 ←(矢印)はメタクラスター1で*(アスタリスク)はメタクラスター12。B)CD16発現強度 C)CD56発現強度。データはKaluzaでコンペンセーションとlogicleスケーリングを済ませた後、Kaluza Cytobank PluginでCytobankへアップロードした。viSNE設定はマニュアルゲーティングで細胞集団を同定するのに用いられた11 マーカー、iterations = 3,000、perplexity = 30、theta =0.5。FlowSOM設定は以下のとおり。Clusering channel = viSNEと同じ、Clustering method = Hierarchical Consensus、Number metaclusters=12、Number clusters = 121。プロットはイメージ目的。
viSNEとFlowSOMの組み合わせはサンプル間の相対的な比較を可能とし、特定のマーカー発現のプロットで表示することで、より直感的に理解できるように可視化します(Figure 5)。CD16+の細胞集団であるメタクラスター1(Figure 5Aの矢印の島)は、sample Gと比較してsample BとFでは顕著に欠損しています。また、sampleFでは他の2つに較べてCD56を発現しているクラスター12(Figure 5Aのアスタリスクの島)の細胞数が増加していることも容易に把握できます。
もうひとつの教師なしアルゴリズムで、類似する細胞をグループ化するツールにSPADE(Spanning-tree Progression Analysis of Density-normalized Events)4 があります。SPADEでは類似するフェノタイプを持つ細胞をグループ化し階層的クラスターにし、 サンプル間の分布の違いを見ることができます(Figure 6)。SPADEによって自動作成されたクラスターを任意の細胞集団としてまとめ、バブルとして表示することもできます。
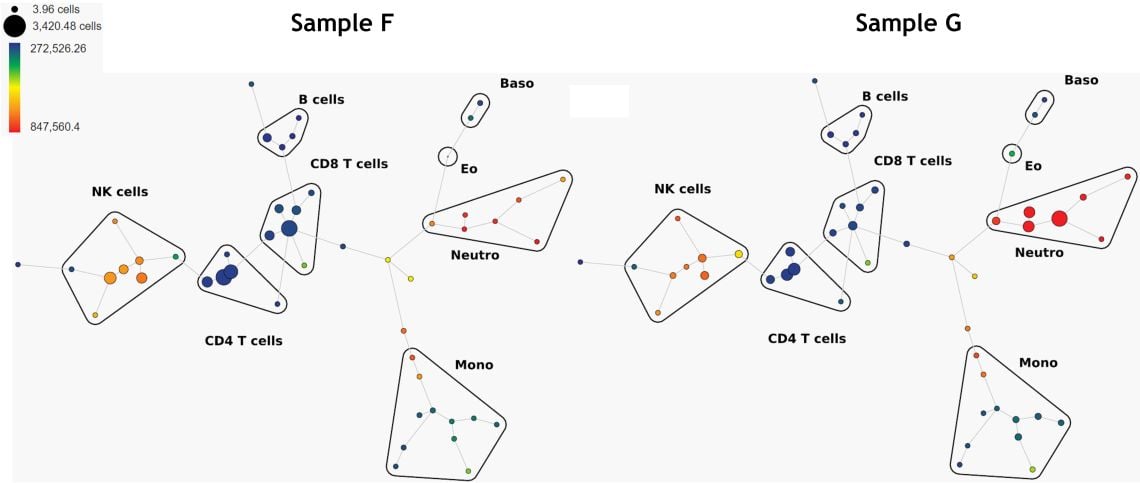
Figure 6. 2サンプルのSPADE結果の比較。クラスターのカラー表示はCD16発現強度(左上のカラーバー)。 データはKaluzaでコンペンセーションとlogicleスケーリングを済ませた後、Kaluza Cytobank PluginでCytobankへアップロードした。SPADE設定はマニュアルゲーティングで細胞集団を同定するのに用いられた11 マーカー、ダウンサンプリング=10 %、50 ノード。プロットはイメージ目的。
それぞれのLeukocyte集団のより詳細な免疫プロファイルは、マニュアルゲーティングを深めることで可能となります。しかしながらこの方法には発現パターンの知見が必ず必要となります。例としてFigure 7にCD4+ T 細胞サブセットを示します。
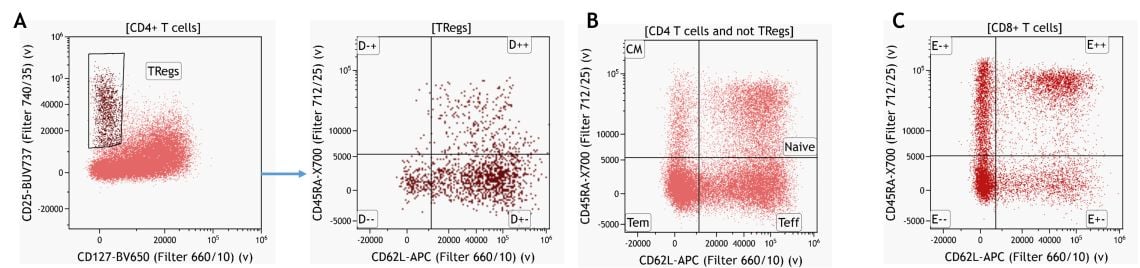
Figure 7. 知見をもとにした制御系T細胞のマニュアルゲーティング例。 A) Treg 細胞のサブセット B)Treg 以外のCD4+T細胞のサブセット C)CD8+T細胞のサブセット。Kaluzaを使用し、プロットはイメージ目的。
サンプル間の比較をするには、比較グラフや統計値を用いたり、オーバーレイ表示やKaluzaのComparison Plotを用います(Figure 8)。これらのアプローチは、違いがありそうだという仮説があるときにはとても有用となります。
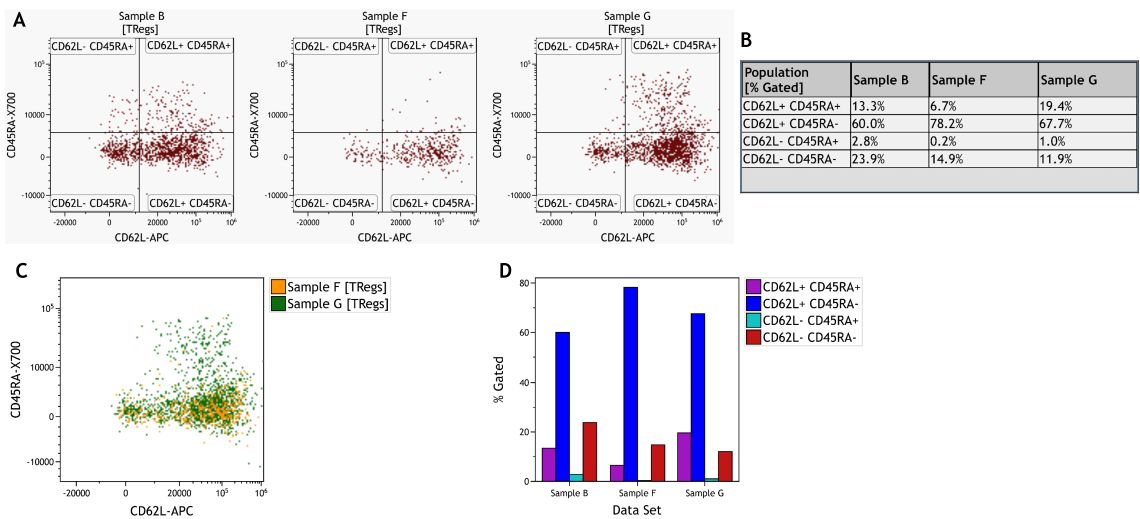
Figure 8. サンプル間のTregサブセット比較。A)各サンプルのドットプロット。B)各サンプルとサブセットの統計値表。C)2つのサンプルのオーバーレイドットプロット。D)各サンプルにおける各サブセットの%グラフ(Kaluza Comarison Plot)。Kaluzaを使用し、プロットはイメージ目的。
教師なしでのT細胞サブセットの同定のため、viSNEを用いた解析を行いました。プレゲートとしてCD3+T細胞までゲーティングで絞り、viSNEの解析対象としました。Figure 9Aに、viSNEマップでのCD4とCD8の発現強度を示します。Figure 7 で示したマニュアルゲーティングと同様に、CD45RAと CD62Lの発現パターンをT細胞全体を対象にクワドラントゲートで同定し、viSNEマップ上に各サブセットをオーバーレイ表示しました(Figure 9B)。最後に、FlowSOMで10メタクラスターへ階層的クラスタリング(Figure 9C)し、マニュアルゲーティングしたもの(9B)と比較すると、ほぼ一致する同定がされていることが明らかとなりました。
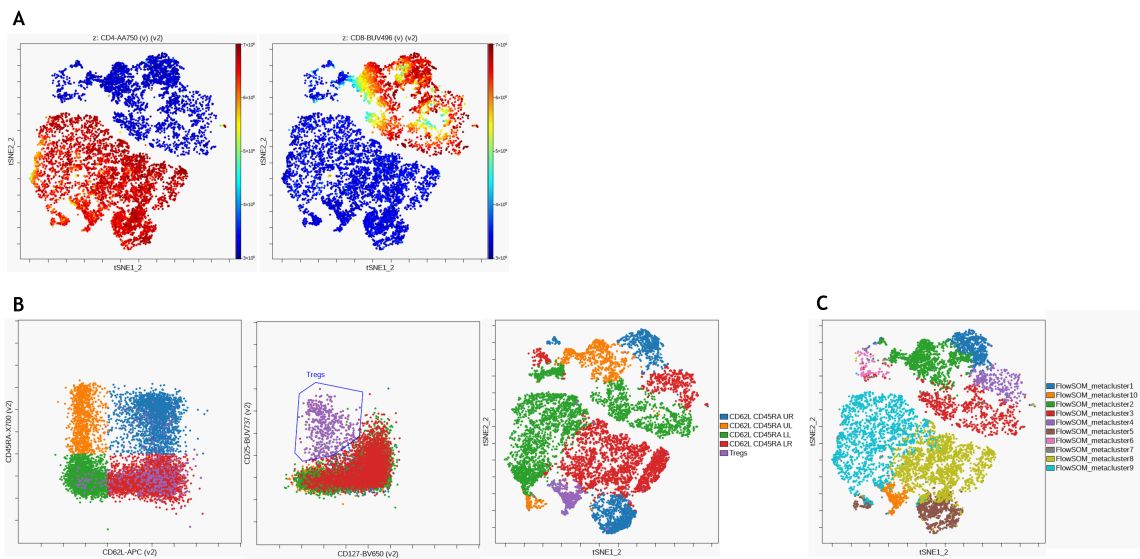
Figure 9. 教師なしアルゴリズムでのT細胞サブセット解析。A)同一viSNEマップでCD4(左)とCD8(右)の発現をカラー表示。下半分の島がCD4+の集団で、右上の島がCD8+の集団であることがわかる。viSNE設定はiteration =2,000、perplexity =50.B)マニュアルゲーティングでのCD62L vs CD45RAプロットでのクアドラントゲーティング(左)、Tregゲーティング(中央)とそれらをviSNEマップ上にオーバーレイ表示(右)。C)FlowSOMによるメタクラスターをviSNEマップ上にオーバーレイ表示。FlowSOM設定は100クラスター、10メタクラスター、ノーマライズ有。データはKaluzaでコンペンセーションとlogicleスケーリングを済ませた後、Kaluza Cytobank PluginでCytobankへアップロードした。プロットは
イメージ目的。
Figure 10では、マニュアルゲーティングとFlowSOM教師なしクラスタリングとのCD8+メモリーサブセットの同定を比較しました。
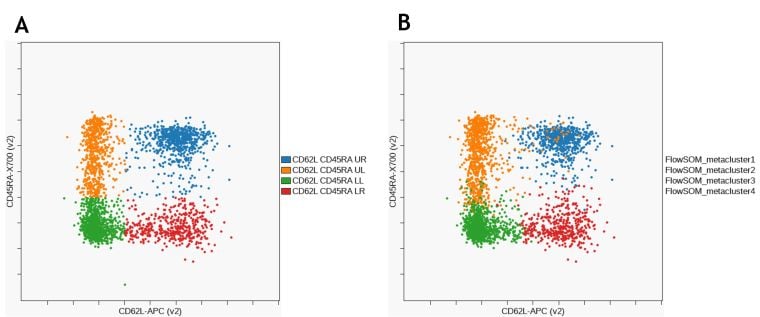
Figure 10. CD8+T細胞メモリーサブセットの同定の比較。A)マニュアルゲーティング、B)FlowSOM。データはKaluzaでコンペンセーションとlogicleスケーリングを済ませた後、Kaluza Cytobank PluginでCytobankへアップロードした。プロットはイメージ目的。
まとめ
3名のドナー血液からの20マーカーパネルサンプルデータをCytoFLEX LXで取得し、マニュアルゲーティングでLeukocyteサブセットを同定した後、T細胞サブセットについての詳細解析を行いました。次に同一のデータについてviSNEを用いて2次元のviSNEマップで見る方法と、viSNEとSPADEを用いたサンプル間の比較について説明しました。最後にFlowSOMによる階層的クラスタリングとマニュアルゲーティングによるサブセット同定を比較しました。
これらviSNE、FlowSOM、SPADEといったマシンラーニングツールは、高次元データの情報を理解または把握しやすくするための可視化を可能とするとともに、細胞サブセットや従来の知見に基づくマニュアルゲーティングでは見落とされていた希少サブセットをバイアスなく同定するのに役立ちます。
Tip
KaluzaとCytobankの操作方法等についてさらに知りたい方は、https://www.beckman.jp/flow-cytometry/software/kaluza またはhttps://www.beckman.jp/landing/japan/2020-cytobank をご覧ください。
本アプリケーションノートは操作方法を案内するものではありません。
テクニカルノートのご用意もあります。お気軽にお問い合わせください。
参考文献
- Amir ED, Davis KL, Tadmor MD, et al. viSNE enables visualization of high dimensional single-cell data and reveals phenotypic heterogeneity of leukemia. Nature Biotechnology. 2013;31(6):545-552. doi:10.1038/nbt.2594.
- Van Gassen S, Callebaut B, Van Helden MJ, et al. FlowSOM: Using self-organizing maps for visualization and interpretation of cytometry data: FlowSOM. Cytometry. 2015;87(7):636-645. doi:10.1002/cyto.a.22625.
- Ortolani C. Antigens. In: Flow Cytometry of Hematological Malignancies. John Wiley & Sons, Ltd; 2011:1-157. doi:10.1002/9781444398069.ch1.
- Qiu P, Simonds EF, Bendall SC, et al. Extracting a cellular hierarchy from high-dimensional cytometry data with SPADE. Nature Biotechnology. 2011;29(10):886-891. doi:10.1038/nbt.1991.
For Research Use Only. Not for use in diagnostic procedures.

