NGS ライブラリ自動調製システム Biomek NGeniuS を用いた Illumina DNA Prep Kit の自動化
はじめに
この数十年間、DNAシーケンシングは、1970年代のフレデリック・サンガーによるチェーンターミネーション法の発見から2005年に導入された大規模並列シーケンシング法へと発展してきました。これらの現代のシーケンシング技術は、次世代シーケンシング(NGS)とも呼ばれ、ラボで一般的に使用される実験手法になっています。NGSワークフローの重要なステップの1つにシーケンシングに対応したDNAフラグメントライブラリの調製がありますが、残念ながら、このプロセスはNGSワークフローにおける課題となっていました。NGS用ライブラリの調製は煩雑で、調製するライブラリの種類によっては2時間半から数日もの時間を要する場合があります。サンプルとアダプタのペアリングを正確に記録するためには、細心の注意を払わなければなりません。各アダプタを手動でピペッティングした場合、エラーが起こりやすく、 正しいアダプタでライブラリを作成できないことがあります。多くのプロセスに時間を要し、セーフストップポイントもないことから、1日の作業時間が極めて長くなってしまいます。こうしたことから、多くのラボでは、NGSのこれらの重要なステップの自動化を求める声が高まってきています1。
IlluminaのDNA Prepライブラリキットは、ライブラリを約3時間半で調製できる方法です。IlluminaのNextera XTとは異なり、DNA Prepは小型ゲノムと大型ゲノムの両方に適し、1 ~ 500 ngのインプットに対応しています。タグメンテーションステップで、トランスポゾンがDNAを切断し、各断片の末端にヌクレオチド断片を挿入します。追加されたDNA断片は、増幅中に付加されるインデックスアダプタの一部と相補的です。この方法は、タグメンテーションだけでなく増幅プロセスもビーズ上で行うことができるという点でも、IlluminaのNextera XTとは異なります。ビーズベースのタグメンテーションでは、ビーズに結合できる断片のサイズを制限することにより、約300 ~ 350塩基対の断片を選択できます。PCR後に両端クリーンアップを行い、不要な断片を除去します。QC(精度管理)ステップでは、1 μLをAgilent Bioanalyzer で分析し、サイズの分布を測定します。プロトコル終了時のライブラリの平均サイズは約600 bpである必要があります2,3。
本アプリケーションノートでは、10 ngと500 ngでBiomek NGeniuSシステムを用いた自動調製を実証し、Biomek NGeniuSシステムで処理したデータと、メーカーが公開しているデータとを比較しました。本アッセイでは、ハンズオンタイムが短縮され、システムのセットアップ時間も短くなっています。

Figure 1. Biomek NGeniuS を用いたIllumina DNA Prep プロトコルのワークフロー。赤色は、Biomek NGeniuS システム上で実施できないプロセスを示します。青色はBiomek NGeniuS システム上で実施可能です。
方法
1. セットアップ
ライブラリ調製前にCoriell NA12878 DNAサンプルを、開始に適した濃度(最大100倍)に希釈します。この値は、ライブラリ調製のインプット量(ng)をインプット容量(μL)で割ることで算出しました。この濃度は、ノーマライゼーションセクション(Figure 1のdsDNAサンプル希釈)でサンプルを希釈した濃度です。最大希釈率の上限は100倍までです。
サンプルの準備完了後、Biomek NGeniuSカスタマーポータルにサンプルをセットします。はじめに、+createボタンを選択してシステムで実行するバッチを作成しました(Figure 2)。次に、Illumina DNA Prep Appを選択します。セットアップは、バッチ情報B(バッチ名と実行するサンプル数)、App セッティング、セクション、サンプルデータの4つのパートに分かれています(Figure 3)。App セッティングにはライブラリキット固有の変数が含まれており、毎回変更することも、ラボの管理者にて設定値を固定することもできます。バッチ名は、処理するサンプル別のランネームを付けます。サンプル数は、入力ボックスの下の灰色の値で示されているように、このアプリケーションでは4 ~ 24までの任意の数を入力します。App セッティングの各設定とその説明はTable 1.をご参照ください。

Figure 2. 上図の +create ボタンを用いて新しいバッチセットアップを開始します。

Figure 3. バッチを実行するためのバッチ情報とApp セッティング
Table 1. App セッティング内の各設定とその説明

次にセクション欄にデータを入力します(Figure 4)。Illumina DNA Prepの場合、4つのセクションが使用されることが想定されます。Figure 3のセクションマーカーの直下で、プロセスのどこから開始するかをユーザが選択することができます。最初のセクションであるノーマライズサンプルを手動で行いたい場合は、Start at sectionを利用してTagment gDNAを選択します。出発点はドロップダウンメニューで選択できます。出発点は、ライブラリ調製キットの使用説明書に規定されたセーフストップを使用しますが、Biomek NGeniuSシステムでのセーフストップにもなります。セクションの左側にある青色のスライダーで、サンプル処理を終了するセーフストップを選択することができます。装置は無人で運転するよう設計されていますが、次の工程で運転を再開する前に、処理を中止してサンプルを安全に保存する方法も選択できます。
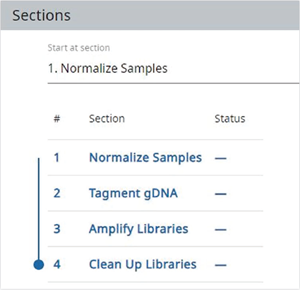
Figure 4. Illumina DNA Prep アプリケーションの場合のセクション

Figure 5. サンプルデータの情報
2. ライブラリー調製
10 ngと500 ngのインプットでBiomek NGeniuSシステムを複数回実行しました。インプットサンプルは、処理前のシステムによるサンプルインプットの希釈率が1:100以内でなければなりません。インプットDNAの総ナノグラム数に基づいて、ライブラリ濃度として使用する標準濃度までサンプル希釈をシステムにて行いました。さまざまな数のサンプルを調製し、さまざまなインプットサンプルの処理をテストしました。処理のために用いた変数と環境条件はTable 2のとおりです。
全ての試薬が正しいストレージに分注された後、試薬を取り出すよう指示が表示され、プロトコル開始時のユーザ入力値に基づいた、ライブラリ調製の推定完了時間が表示されます。サンプル処理記載のプロトコルからの唯一の例外として、ビーズ洗浄に要する時間を短縮するために、プロトコルに記載された200 μL全量ではなく、50 μLのエタノールを用いてビーズうを洗浄しました。運転完了後、サンプルは自動的にAgilent Bioanalyzerにて分析されます。サンプル収量の決定にはThermo Qubit高感度アッセイを使用しました(Table 3)。シーケンシングは、Illumina MiSeq 2x 151 cycle v2ケミストリーキットに、10 pMのマルチプレックスサンプルを用いて行いました(10 ng)。データはIllumina BaseSpaceで解析しました。BaseSpace(v1.1.4)のBWA Alignerアプリケーションを用いてヒトレファレンスゲノム(UCSC HG19)のデフォルト設定にリードをアラインすることで、ライブラリを解析しました。
Table 2. Biomek NGeniuS システムで処理したサンプルのサンプル情報、処理情報、環境条件

結果・考察
Agilent Bioanalyzerで得られた結果を、メーカーが公表している結果と比較しました。
Biomek NGeniuSシステムで調製したライブラリはメーカーの推奨範囲内であり、サンプル調製に成功したことが確認できました(Figure 6.とFigure 7.はBiomek NGeniuSシステムで異なる実行を2回実施した結果です。Illumina DNA Prepキットの典型的な平均ライブラリサイズは、Agilent Bioanalyzerを使用した場合600塩基対です3)。Thermo Qubit高感度アッセイの結果をTable 3に示します。

Figure 6. Biomek NGeniuSシステムで調整したライブラリのPCR後のバイオアナライザー分析結果(500 ngのライブラリ調製)

Figure 7. Biomek NGeniuSシステムで調製したサンプルライブラリのPCR後のバイオアナライザー分析結果(10 ngのライブラリ調製)
Table 3. サンプルインプット10 ngのライブラリ調製におけるQubit収量と平均サイズ

Biomek NGeniuSシステム調製からのシーケンシングにより、11,718,608のRAWリードと6,148,202のパスフィルターリードが生成されました(52.4%)。Q30スコアは総収量965.27 Mbに対して90.12 %を上回りました。パスフィルターリードの94%が同定に成功し、11個のライブラリ全てを示します(Table 4)。シーケンシングの成功は、アライメントとリードの高いペアリング率および低い重複率によって示されます(Illumina.com)。全てのライブラリでアライメントリードは99%を超え、適切にペアリングされたリードは75%を超え、重複は6%未満であったことから、サンプル調製とシーケンシングに成功したことが確認できました。
Table 4. 10 ngでライブラリを調製した場合のBaseSpace解析によるシーケンシングリード、アライメント、重複、適切にペアリングされた割合の結果

まとめ
Biomek NGeniuSシステムの試作機を用いてIllumina DNA Prepライブラリキットを自動化することにより、収量、サイズおよびシーケンシングデータから、アライメントが99%を超え、重複率が低く、適切にペアリングされた断片の割合が75%を超えるライブラリが得られることが示されました。より一貫性のある断片化サイズの取得、サンプル間のばらつきを抑え、ライブラリ調製の一貫性を向上させるために、断片化における混合をより改善するさらなる最適化を試みています。
使用した試薬・消耗品・機器など
Table 5. Illumina DNA Prep用サンプルの調製に用いたサンプルの種類

Table 6. Illumina DNA Prepキットを用いたライブラリの調製とIlluminaシーケンサーに用いた試薬

|
Table 7. Illumina DNA Prepのサンプル調製・処理に用いた機器
|
Table 8. サンプル処理に必要な消耗品
|
参考文献
- Bruinsma S, Burgess J, Schlingman D, Czyz A, Morrell N, Ballenger C, Meinholz H, Brady L, Khanna A, Freeberg L, Jackson RG, Mathonet P, Verity SC, Slatter AF, Golshani R, Grunenwald H, Schroth GP, Gormley NA. Bead-linked transposomes enable a normalization-free workflow for NGS library preparation. BMC Genomics. 2018 Oct 1;19(1):722. doi: 10.1186/s12864-018-5096-9. PMID: 30285621; PMCID: PMC6167868.
- Illumina DNA Prep Reference Guide Document 10000000025416 V09, June 2020, https://sapac.support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/illumina_prep/illumina-dna-prep-referenceguide-1000000025416-09.pdf
- Illumina Nextera DNA Flex Library Preparation Kit Technical Data Sheet, https://www.illumina.com/content/dam/illuminamarketing/documents/products/datasheets/nextera-dna-flex-data-sheet-770-2017-011.pdf
重要:本アプリケーションテンプレートにおいて、ベックマン・コールターは、明示または黙示を問わず、特定の目的や商品性への適合性の保証、本アプリケーションテンプレートが他者の権利を侵害していないことの保証を含め、いかなる種類の保証も行いません。全ての保証は明示的に放棄されます。本アプリケーションテンプレートはお客様ご自身のリスクのみにおいて使用されるものとし、ベックマン・コールターは一切の責を負いません。本アプリケーションテンプレートは、アプリケーションが選択・作成された時点で示されていたケミストリーキットのバージョンとリリース日についてBiomek NGeniuSシステムでの使用を確認したものですが、ベックマン・コールターは疾患やその他の臨床状態の診断への使用についての検証は行っていません。製品および本書に示したアプリケーションは、診断手順での使用を意図しておらず、検証も行っていません。開発段階では性能特性は検証していません。
Beckman Coulter およびBeckman Coulter ロゴは、Beckman Coulter, Inc. の登録商標です。
| PDFダウンロードはこちら > > Click here! |
|
|
NGSライブラリ自動調製システム Biomek NGeniuS は、次世代シーケンサー用ライブラリ調製の自動化システムです。煩雑で時間の掛かるNGSのライブラリ調製を、少ない作業時間、高い柔軟性を持つシステムで運用したい研究室に適しています。ユーザーフレンドリーなソフトウエアは高度なプログラミングスキルを必要としないため、簡単に誰でもセットアップできます。 |




