ナノフローサイトメーターCytoFLEX nano: FCMPASS™を用いた散乱光データの直径への変換
目的
本稿では、FCMPASS™ソフトウエアを使用して、ナノフローサイトメーターCytoFLEX nanoで取得したバイオレット側方散乱光(VSSC)データを、直径という標準単位(ナノメートル(nm))に変換する方法を説明します。散乱光データを直径に変換する最大の目的は、異なる原理の測定装置間でも比較を可能にすることです。もう一つの利点として、その特性を評価することによって生物学的洞察を得ることが挙げられます。
このアプリケーションノートでは、以下について説明します。
-
ナノ粒子研究において、測定原理による任意の単位を標準単位(直径: nm)に変換することの重要性。
-
近似屈折率(RI)、粒径およびRIが既知のキャリブレーション標準粒子、Mieモデルを用いた他社製ソフトウエアFCMPASS™を使用して、CytoFLEX nanoのVSSC1パラメータデータを直径の標準単位であるnmに変換する方法。
-
FCMPASS™単位変換したFCSファイルを、CytExpert nanoソフトウエアにインポートする方法。
注意
Flow cytometry post-acquisition analysis(FCMPASS™)は、任意の単位で表される散乱光データをナノメートル(nm)単位に変換するためのソフトウエアです。その方法を本アプリケーションノートで説明しますが、この方法は、フローサイトメトリーデータからナノ粒子の直径を推定する方法の1つです。ユーザーのニーズや実験条件に最適な変換方法を使用することを推奨します。
FCMPASS™は他社製ソフトウエアであり、ベックマン・コールターの製品ではありません。このアプリケーションノートでは、CytExpert nanoソフトウエアの有するFCSファイルのエクスポートおよびインポート機能と、FCMPASS™による生体ナノ粒子のサイズ変換について記したものです。ベックマン・コールターはフローサイトメトリーのデータをサイズに変換する際に、特定の方法を支持しているわけではなく、本稿の方法はあくまで一例として示すもので、ベックマン・コールターによって検証および妥当性確認がされたものではありません。
はじめに
ナノフローサイトメーターCytoFLEX nanoは、細胞外小胞(EV)や脂質ナノ粒子(LNP)などを単一ナノ粒子でフェノタイプ決定や特性評価できる先進的なナノ粒子フローサイトメーターです。CytoFLEX nanoは、VSSC2チャネルを用いて粒子の検出ダイナミックレンジを最大1000 nmまで維持しながら、VSSC1検出器の感度を40 nmの粒子(ポリスチレンRI)の検出が可能なレベルまで高めたことでその性能を実現しました。クロスプラットフォームでの比較を可能にするため、当社では、フローサイトメトリーで得た散乱光データをMieモデルソフトウエアを用いて標準単位(nm)に変換することを推奨しています。これは、ISEV-ISAC-ISTH(International Society of Extracellular Vesicles:国際細胞外小胞学会、International Society for Advancement of Cytometry:国際サイトメトリー学会、International Society of Thrombosisand Haemostasis:国際血栓止血学会)が承認し、さらにMISEVガイドラインでISEVが推奨するMIFlowCyt-EVガイドライン(Welsh et al., 2020)を遵守することができます。
FCMPASS™は、ナノ粒子フローサイトメトリーデータの蛍光および散乱光キャリブレーション、最適化、そしてレポート作成を行うための他社製ソフトウエアです(Cook et al, 2023)。自動レポート作成、半自動化された標準物質のクロスキャリブレーションと検出器設定の最適化、およびFCSファイルキャリブレーションのための標準物質のカタログ化を機能として有しています。FCSファイルキャリブレーション時にはMIFlowCytとMIFlowCyt-EVに則った品質管理関連の数値が自動生成されるため、研究者は業界の最も高い基準に則した透明性でデータ報告を行うことが可能です。
ナノ粒子の特性評価において、サイズを知ることは重要です。FCMPASS™は、蛍光および散乱光データに基づいてサイズを算出する方法のどちらにも対応しています。このアプリケーションノートでは、散乱光データを用いたサイズ算出について説明します。FCMPASS™はMieモデルと、サイズと屈折率(RI)が既知のキャリブレーション標準物質を用いてフローサイトメーターの側方散乱光収集角度を導出します。収集角度が導出されると、組成やRIが異なる粒子のサイズと散乱光シグナルの非線形関係をマッピングするためのルックアップテーブルを生成することができます。これらを用いてサイズを推定します(Cook et al., 2020, Welsh et al., 2020, Welsh et al., 2019)。
導出される収集角度の正確性は、使用するキャリブレーション用標準物質の確度に依存します。このため、直径とRIについて十分な特性評価が行われている米国国立標準技術研究所(NIST)の認証を受けたトレーサブルな標準物質を使用します。さらに、ソフトウエアがフローサイトメーターの収集半角(ハーフアングル)を正確に導き出すには、少なくとも4サイズ(80 nm以上で、かつスパンが200 nm以上)のNISTトレーサブルポリスチレン標準粒子が必要です(Welsh et al,. 2020)。このアプリケーションノートに記載されているビーズサイズで、このMie散乱領域をカバーします。
粒子の散乱光データをナノメートルに変換するために、もう一つ重要なステップを追加する必要があります。それは変換対象粒子のおおよそのRIを得ることです。FCMPASS™は該当するEVのRI範囲をデフォルト値として提供しますが、ユーザーは必要に応じてコアシェルモデルを微調整することもできます。以下にワークフローをステップバイステップで例示します。
ハーフアングルの同定
-
CytExpert nano:標準粒子を用いたデータの取得
-
FCMPASS™:標準粒子のVSSC2データからハーフアングルを近似
-
ハーフアングルとデータキャリブレーションの確認
-
FCMPASS™:VSSC2で得た近似ハーフアングルを用いたVSSC1データのキャリブレーション
-
CytExpert nano:キャリブレーション済みCytoFLEX nano.FCSファイルのインポート
CytExpert nano:VSSC1チャネルでのEVサンプルと標準粒子のデータ取得
Note: VSSC1チャネルは感度が非常に高いため、このチャネルで検出可能な最大の粒子であってもFCMPASSでハーフアングルを近似するための基準(80 nm以上かつスパンが200 nm以上の、少なくとも4サイズのNISTトレーサブル標準粒子)を満たすには小さすぎます。しかし、VSSC1とVSSC2検出器は同一のハーフアングルであるので、VSSC1より感度の低いVSSC2検出器を使用して、VSSC1とVSSC2パラメータ両方のハーフアングルを導出することができます。
この方法についての詳細は、メーカーが提供している文書https://resources.fcmpass.com/examples/known-anglescatter-calibrationをご参照ください。
用意するもの
-
CytExpert nano(ベックマン・コールター、ソフトウエア ver.1.2.0.34以上)
-
FCMPASS(FSMpass社、ソフトウエア ver.5.0.7以上)
-
CytoFLEX nano Multi-intensity Fluorospheres(ベックマン・コールター、製品番号:C92889)
-
NISTトレーサブル標準粒子(40 nm、80 nm、125 nm、200 nm、300 nm、450 nm)入りデモキット
-
超純水またはHPLCグレード水(Thermo Fisher、製品番号:W8-1)
-
PBS(Cellarcus、製品番号:CBS4B-100 ml)
-
20 nmフィルター(Cytiva、製品番号:6809-2002)
-
12x75mm ホウケイ酸ガラスチューブ(VWR 製品番号:47729-570)
方法
CytExpert nano:標準粒子を使用したデータ取得
装置の起動とDaily QC
-
CytoFLEX nanoの電源を入れ、CytExpert nanoソフトウエアを開きます。
-
「System Startup」でCytoFLEX nanoを起動します。
-
CytoFLEX nano QC ScatterspheresおよびQC Fluorospheresを使用してDaily QCを実行します。「CytoFLEX nano Instructions for Use(CytoFLEX nano 取扱説明書):IFU」を参照のうえ、QCプロンプトに従って実行します。不具合が発生した場合は、CytoFLEX nano IFUを参照してQCを再テストします。
-
CytoFLEX nano Multi-intensity Fluorospheresを使用してSensitivity Monitorを実行します。不具合が発生した場合は、CytoFLEX nano のIFUを参照し、Sensitivity Monitorを再テストします。
-
On-board Cleanを実行します。
-
Baseline monitorを実行して、装置が清浄であることを確認します。CytoFLEX nano IFUのSection 6~44をご参照ください。
-
20 nmシリンジフィルターでHPLCグレード水をろ過します。
-
デモキットのストックビーズチューブ(80 nm、125 nm、200 nm、300 nm、450 nm)に750 μLのろ過済みHPLCグレード水をそれぞれ添加します。
-
すすいだ12x75 mmホウケイ酸チューブ5本に、以下のようにラベル付けをします。
-
各チューブに150 μLのろ過済みHPLCグレード水を加え、対応するチューブに50 μLの各ビーズを加えます。
-
New experimentを作成します。
-
VSSC1およびVSSC2ヒストグラムを作成します。VSSC1-AとVSSC1-H、VSSC2-AとVSSC2-Hの二変数プロットを作成します(Figure 1)。
-
「Acquisition Settings」を開きます。「QC Gain」をクリックし、その日合格したQC結果からQC Gainを適用します。「Trigger」をVSSC1に、「Threshold」を200に設定します(Figure 2)。
-
「Acquisition」パネルで、「Events to Record」のチェックを外します。「Time to Record」を「30 seconds」、「Flow Rate」を「Slow」(1 μL/min)に設定します。
-
Tube 1に「Water」とラベルします。
-
ろ過済みHPLCグレード水のチューブをチューブホルダーにセットし、「Run」を選択します。VSSC1-Hの水のノイズが、Figure 3の左図のように単一の鋭いピークであることを確認します。装置または水サンプルチューブが清浄でなかった場合は、すすいだチューブに新たにろ過済みHPLCグレード水を入れたものを用意します。ノイズが高いままの場合は、Onboard Cleanを実行し、Baseline monitorをチェックします。
-
ろ過済み水のチューブの測定をする際は、イベントレートが50-100 Events/Sec (EPS)の範囲になるようにthreshold を調整します。これが、現在のQCゲイン設定での実験のthresholdになります。
-
「Record」をクリックしてデータを取得します。
-
データ取得が終了したら、「Unload」をクリックします。
-
Waterチューブを選択し、Add Tube
 を5回クリックしてチューブを5本追加します。これで、全てのチューブに同じQC設定とthresholdが適用されます。
を5回クリックしてチューブを5本追加します。これで、全てのチューブに同じQC設定とthresholdが適用されます。 -
チューブの名前を「80 nm」、「125 nm」、「200 nm」、「300 nm」、「450 nm」に変更します。
-
80 nmサンプルチューブをチューブホルダーにロードします。
-
「Record」をクリックしてデータを取得します。
-
データ取得が終了したら、「Unload」をクリックします。
-
手順22~24を残りの全てのチューブで繰り返し、ビーズサイズに対応するチューブをロードします。各標準ビーズで典型的に認められるピークのヒストグラムをFigure 4に示します。
-
以下のようにゲートを描画します。
-
VSSC2-Hヒストグラムのポピュレーションの周囲にゲートをタイトに描き、「V2」とラベルします(Figure 8)。
-
CytExpert nanoウィンドウの上部にあるツールバーの
 アイコンをクリックして統計表を作成します。新規に作成されたテーブル上で右クリックし、「Statistics Setting」をクリックします(Figure 9)。
アイコンをクリックして統計表を作成します。新規に作成されたテーブル上で右クリックし、「Statistics Setting」をクリックします(Figure 9)。 -
「Statistics Setting」ウィンドウの「Statistics」タブで、VSSC2-Hの「Median」と「CV」のチェックボックスにチェックを入れます。「Events」、「% Total」、「% Parent」のチェックを外します。一番下は「All Tubes」に変更します(Figure10)。
-
「Population」タブで、「V2」以外の全ゲートのチェックを外します。「All Tubes」を選択し、「OK」をクリックします(Figure 11)。
-
各ビーズサンプルの「V2 Singlets」と「V2」ゲートを適宜調整します。80 nm、125 nm、200 nm、300 nm、450 nm標準PSビーズのVSSC2-H Medianを記録します(Figure 12)。
-
以下のリンクからFCMPASSソフトウエアをダウンロードし、インストールします。
https://www.fcmpass.com/download Download | Home (fcmpass.com). -
Windowsツールバーの検索機能を使用してFCMPASSを検索します。FCMPASSソフトウエアのアイコン「
 」をダブルクリックして、ソフトウエアがロードされるのを待ちます。FCMPASSを初めて使用する場合は、ナノフローサイトメーターCytoFLEX nanoに付属のインダストリアルライセンスを使用してFCMPASSアカウントを作成・登録してください。
」をダブルクリックして、ソフトウエアがロードされるのを待ちます。FCMPASSを初めて使用する場合は、ナノフローサイトメーターCytoFLEX nanoに付属のインダストリアルライセンスを使用してFCMPASSアカウントを作成・登録してください。 -
VSSC2データを用いたハーフアングルの近似を始める前に、ビーズとCytoFLEX nanoの固有情報と仕様をFCMPASSにカタログ化する必要があります。
-
トレーサビリティと正確な計算を確保するため、フローサイトメーターとビーズに関する正しい情報をソフトウエアカタログに入力することが重要です。
-
FCMPASSのメインウィンドウで「Catalogue」をクリックします(Figure 13)。
-
カタログにフローサイトメーターを追加します。「Cytometers」タブの適切なフィールドに、フローサイトメーターに関する情報を入力します(Figure 14)。
1. Cytometer ID: フローサイトメーターの名称
2. Manufacturer: フローサイトメーターのメーカー
3. Model: フローサイトメーターのモデルまたはバージョン
4. Import Reference File: FCSファイルからキーワードを追加
-
「Import Reference File」(4、Figure 14)をクリックします。開いたウィンドウで、フローサイトメーターに対応するデータファイルの場所に移動します。任意のファイルを選択します。この操作で、フローサイトメーターのシリアル番号が自動的にインポートされます。
-
「Add Cytometer」をクリックして装置情報を保存します。「Add Cytometer」の位置は、Figure 15 をご参照ください。
-
これで、このフローサイトメーターは「Selected Cytometer」ドロップダウンメニューに表示されます(Figure 15)。
-
「Light Scatter」タブをクリックし、NISTトレーサブルビーズの情報を入力します。
-
CytoFLEX nanoキャリブレーションPSビーズの分析証明書(CoA)を準備します。
-
カタログに散乱光ビーズを追加します。「Light Scatter」タブで、NIST Sizingビーズなど、サイズ計測に使用するビーズの情報を入力します。この情報はメーカーが提供するビーズのCoAに含まれます(Figure 16)。
1. Diameter(nm): ビーズの直径(サイズ)。名前やサイズのカテゴリではなく、メーカーが測定した平均直径を入力します。
2. Diameter (CV%): CV値を%なしで入力します(2.0 %は2.0と入力)。CVがメーカーから提供されていない場合、標準偏差を平均直径で割れば求められます。
3. Refractory Index (RI): メーカーから提供されたRIを入力します。
4. Manufacturer : ビーズのメーカー名。
5. Lot Number : CoAに記載されているビーズのロット番号。
6. RI Measurement Wavelength (nm): 上記のRIを測定した波長を入力します。
7. Composition : ビーズの原材料の種類:ポリスチレン、シリカ、その他。材料が異なるとRI値も異なるため、区別することが重要です。
8. Catalogue Number : メーカーから提供された製品番号。
-
全てのビーズを入力したら、ビーズセットを作成してビーズをグループ化します。ビーズセットを作成するには、「Selection」列で、新規ビーズセットに追加するビーズを選択します。次に、「Create Set」をクリックします(Figure17)。
-
このキャリブレーションに使用するNISTトレーサブル標準粒子に対応する全てのビーズのボックスにチェックを入れます。「Create Set」をクリックし、プロンプトが表示されたらビーズセット名に「CytoFLEX nano Calibration PS beads」と入力します(Figure 18)。新しいビーズセットが「Display Set」ドロップダウンメニューに追加されます。
-
FCMPASS Catalogueウィンドウ右上の「X」ボタンをクリックし、カタログを閉じます。
-
メインメニュー(Figure 13)に戻り、「Experiment Calibration」をクリックして、VSSC2データを用いたハーフアングルの近似を開始します。
-
キャリブレーションするフローサイトメーターを選択してデータセットを作成します。次に「+」アイコンをクリックします。以下の情報を入力し、完了したら「Save」をクリックします(Figure 19)。
-
新しく作成したデータセットを選択し、「Begin Calibration」をクリックします。
-
「File Import」で「+」アイコンをクリックしてディレクトリを呼び出し、キャリブレーションするFCSファイルを追加します。CytExpert nano.fscファイルは、「Documents」フォルダの「CytExpert nano Data」フォルダ内の「Experiment」フォルダにあります。キャリブレーション後、レポートとキャリブレーション済みデータの入った新しいフォルダが、元のFCSファイルと同じサブフォルダに作成されます。ここで、キャリブレーション用としてゲインとトリガー設定が同じデータサンプルを選択することができます。キャリブレーションをさらに検証するため、Medianの収集を行ったビーズのデータファイルを追加することもできます。ファイルには、そのサンプルの目的に応じて、ラベル、サンプル、FL Calibrator、またはSSC Calibratorを加えることもできます(Figure 20)。
-
「Next」をクリックします。もう一度「Next」をクリックして「Fluorescence Calibration」をスキップし、「Scatter Calibration」に進みます。
-
「+」記号をクリックして、ハーフアングルの近似に使用するチャネルを表示します。VSSC2-Hを選択します。VSSC2-HのMedianを得るために使用した試薬セットに、ビーズセットCytoFLEX nano Calibration PS Beadsを選択します。
-
対応するビーズのVSSC2 Medianデータを「Acquired Stat(au)」列に入力します。CVデータを「Acquired CV (%)」列に入力する必要はありません。空のセルは削除・非表示になります。チャートをリセットするには、必要に応じて「Reset Inputs」をクリックします。完了したら、「Next」をクリックします(Figure 21)。
-
準備ができたら、「Calibrate」をクリックします。キャリブレーションが成功すると、キャリブレーションされたファイルのリストとともに確認プロンプトが表示されます(Figure 22)。
-
FCMPASSは、FCMPASS Figure、Encrypted_Report.exRNA、FCMPASS Report.xlsxとともに、キャリブレーションデータの入ったフォルダを新規ウィンドウで開きます。新規フォルダは、手順50で選択したフォルダ内にあります(Figure 23)。
-
「FCMPASS Figure1.png」とラベルされたpngファイルを開いて確認します。
-
Plot A(Figure 24およびFigure 25の左上図)で、Closest fitting half-angleを特定します。これはFCMPASSがVSSC2のMedianから近似したハーフアングル値です。closest fitting half-angle(曲線のピーク)に対応する「% Fit Confidence」を確認します。Fit Confidenceが90%以上であれば、そのハーフアングルは問題ありません。「% Fit Confidence」のピークが90%未満の場合は、Daily QCが合格であったか、ピークデータのCoAと取得したMedianが正しく入力されているかを確認します。Figure 25 に結果を示します。
-
次に、「Plot B Model Fit」で、取得したMedianデータが想定した散乱断面積に適合するかを確認します。標準粒子が相関関係と一致しない場合は、入力したMedianが正しいか確認します。Figure 24の右上図をご参照ください。
-
次に、「Plot C Model Fit」で、取得したビーズサイズのMedianデータが予測値と同等かを確認します(Figure 26)。「Poor Fit」や「Inaccurate」領域に粒子が見つかった場合は、情報(MedianおよびCoA)が正しく入力されているか確認します。キットに入っていたもの以外の粒子を使用する場合は、キャリブレーション済みの粒子サイズ測定装置で粒子径を検証します。その粒子が確度不良と判明した場合、同じサイズの代替粒子を探します。場合によっては、メーカーから提供されるデータや組成に誤りがあり、ハーフアングルの近似が正しくない値になる可能性があります。
-
Plot D(Figure 27)は、装置のRIに関する散乱性能プロファイルです。Plot A、B、およびまたはCが一致しない場合は、データが正確に入力されているか確認します。データが正確でないと、ハーフアングルの同定やその後のキャリブレーションの正確性に影響が及ぶためです。
-
次にVSSC1散乱光チャネルでキャリブレーションするためのハーフアングルの近似値を記録します。CytExpert nanoソフトウエアに戻ります。
-
サンプル測定前に、以下の基準を満たしていることを確認します。
-
手順62の条件を満たす場合、CytoFLEX nano IFUで推奨されているコントロールとサンプルの測定を開始します。
-
実験サンプルとコントロールチューブのゲイン設定とを同じにして、CytoFLEX nano Calibration PSビーズセットの80 nmと125 nmの標準ビーズを測定します。複数のゲイン設定を使用する場合は、使用した全てのゲイン設定について80 nmおよび125 nm標準ビーズの測定を繰り返します。
-
FCMPASSソフトウエアに戻るか開き、「Experiment Calibration」をクリックします。
-
キャリブレーションするフローサイトメーターを選択して、新しいデータセットを作成します。次に「+」アイコンをクリックします。以下の情報を入力し、完了したら「Save」をクリックします。
-
新規作成したデータセットを選択し、「Begin Calibration」をクリックします。
-
「File Import」で、「+」アイコンをクリックしてディレクトリを呼び出し、キャリブレーションするFCSファイルを選択します(手順64の80 nm、125 nm参照ビーズを含む)。
-
「Next」をクリックします。もう一度「Next」をクリックして「Fluorescence Calibration」をスキップし、「Scatter Calibration」に進みます。
-
「 +」をクリックして、VSSC1キャリブレーションチャネルを表示します。VSSC1-Hを選択し、Bead setにCytoFLEX nano Calibration PS Beadsを選択します。
-
手順64で得た80 nmおよび125 nm NIST標準粒子のVSSC1 Medianデータを「Acquired Stat(au)」列に入力します。キャリブレーション標準粒子のゲイン設定が、キャリブレーションするサンプルと同じであることを確認します。複数のゲイン設定を使用する場合は、使用するゲインごとに別々のキャリブレーションを作成します。
-
ウィンドウ下部の「Advanced Settings」をクリックすると、新規ウィンドウが表示されます。「ModelingParameters」タブを選択します。「Determine Half-Angle」列に既知のハーフアングルを入力できるよう、「DetermineHalf-Angle」をオフにします(Figure 28 左図の青で囲ったボックス)。「Theta」、「Phi」、「Epsilon」という見出しの新しい列が表示されます。ThetaとPhiはフローセルの方向角を表します。Epsilonはハーフアングルです。「Theta」と「Phi」の両方に「90」と入力します。「Epsilon」列の下に手順57の近似したハーフアングルを入力します(Figure 28 右図の青で囲ったボックス)
-
「Particle Modelling」タブに戻ります。データキャリブレーション用の新規チャネルを作成するためにソフトウエアに組み込まれた理論値のCore-shell Parameter RIが表形式で表示されます(Figure 29)。
-
「+」をクリックすると、「Solid Sphere RI Parameters」を追加できます。
-
「Advanced Settings」ウィンドウを閉じると、入力したパラメータが保存されます。
-
準備ができたら、「Calibrate」をクリックします。キャリブレーションが成功すると、キャリブレーション済みファイルの一覧とともに確認プロンプトが表示されます。
-
キャリブレーションが完了すると、キャリブレーション済みデータ、FCMPASS Figure、Encrypted_FCMPASS_Report.exRNA、FCMPASS Report.xlsxの入ったフォルダが自動的に開きます。
-
「FCMPASS Figure1.png」を開いて確認します。キャリブレーションに既知のハーフアングルが使用されているため、Plot Aは表示されません。入力したMedianが、Plots B、C、Dのモデルに適切にフィットしているか確認します(Figure 30)。
-
キャリブレーション済みのCytoFLEX nanoのFCSファイルには、ファイル名の末尾に「FCMPASS」 が付きます。FCS ファイルは既存または新規のExperimentにインポートできます(Figure 31)。
-
ヒストグラムまたは二変数プロットを作成します。プロットのX軸のパラメータラベル上で右クリックします。サンプルのCore-shellとSolid SpheresのRI(デフォルトおよびカスタム)の新しいチャネルパラメータが表示されます。加えて、解析にはCytoFLEX nanoの散乱断面積(Scattering CS)も利用できます(Figure 32)。サンプルデータが正しく表示されるよう、「Plot Settings」は「Linear」に設定し、最小軸値と最大軸値を調整することを推奨します。
試薬の調製
Note: ろ過した水の最初の2~3 mLには製造時の残留粒子が含まれている可能性があるため、必ず廃棄します。その後、新しいガラスチューブに新たにろ過した水を集めます。ガラスチューブは、チューブの内壁を約1 mLのろ過水で洗い流し、洗浄後にデカントして洗浄します。
a. 80 nm
b. 125 nm
c. 200 nm
d. 300 nm
e. 450 nm
Note: 各ビーズの最終濃度は2.5 x 107 particles/mLです。
データの取得
Note: ユーザーはアプリケーションに適したゲインを設定しておき、全てのQC Gainをアプリケーションに適したゲインに置き換えることもできます。
Note: 清浄なろ過済み水サンプルを清浄なCytoFLEX nanoで測定すると、イベントレートが50-100 EPSに調整されている間、シャープなノイズピークが維持されます。装置や水サンプルが清浄でない場合、ノイズピークの鋭さが失われ、残留ノイズ粒子が検出されます。「Baseline Monitor settings」で「Sample Tube」を選択してろ過済み水サンプルの測定を実行すれば、類似のバッファに対して設定されたユーザー定義の合格基準でベースラインモニタリングを実行することも可能です。詳細は、https://www.beckman.jp/en/resources/reading-material/application-notes/cytoflex-nano-flow-cytometer-the-new-approach-to-nanoscale-flow-cytometryをご参照ください。
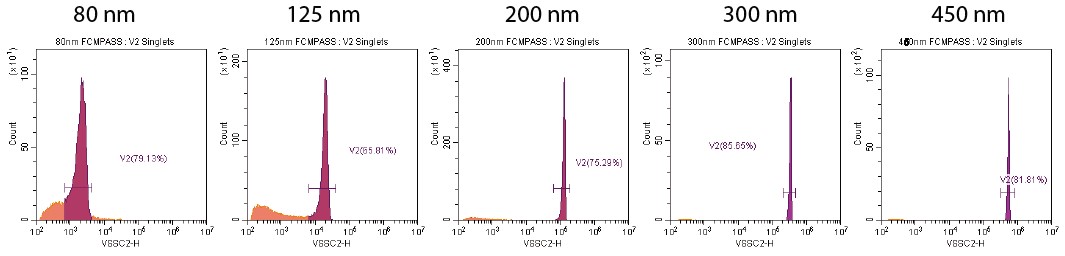
Figure 4. VSSC2でハーフアングルの近似に使用する80 nm、125 nm、200 nm、300 nm、450 nm(左から右)ビーズのVSSC2上でのシグナル。各ビーズはそれぞれのシングレットポピュレーションでゲート。
a. VSSC1-H vs VSSC1-Aプロットで、シングレットポピュレーションの周りにゲートを描き、「V1 Singlets」とラベルします(Figure 5)。
b. VSSC2-H vs VSSC2-Aプロットで、シングレットポピュレーションの周りにゲートを描き、「V2 Singlets」とラベルします(Figure 6)。
c. VSSC1-Hヒストグラムを「V1 Singlets」ゲートで展開し、VSSC2-Hヒストグラムを「V2 Singlets」ゲートで展開します。Figure 7にゲート結果の例を示します。
FCMPASS: 標準粒子のVSSC2データからのハーフアングルの近似
Note: CytoFLEX nanoワークステーションにFCMPASSソフトウエアをインストールしてください。
Note: ビーズ情報を修正するには、修正するビーズの「Selection」列のチェックマークボックスをクリックします。現在のビーズ情報が表示されます。表示された情報を編集して修正したら、「Append Bead」をクリックします。
すでにロードされているExample Setがある場合、このExample Setをキャリブレーションに使用できます。全てのセットに別のサイズ計測ビーズを追加することができます。Example Setを使用する場合は、必ずロット番号などの情報を更新します。ビーズの情報はCoAに記載されています。
a. データ取得日
b. データセット名
c. データセットに関するメモ
Note: 問題がある場合は、入力したMedianのデータに誤りがないか確認してください。
a. FCMPASS Figure 1.png:ハーフアングルと直径のレビュー(モデル適合性と確度の評価も含む)。
b. Encrypted_FCMPASS_Report.exRNA:exRNAデータベースのトレース可能なレポート。
c. FCMPASS Report.xlsx:使用するビーズと装置の全情報と入力値を格納。データのレポート作成ガイドも提供。
Note: ハーフアングルの近似の「Fit Confidence」が90%未満の場合は、原因を特定する必要があります。使用したポリスチレンビーズサイズはMie領域の範囲をカバーすることが試験で確認されているため、問題の原因は、入力された情報が不正確である可能性が高いです。さらに、ハーフアングルの近似を行うチャネルがVSSC2に設定されていることを確認します。CoAをチェックし、手順58、59、および60で概説されている以下のプロットを見直し、ビーズサイズおよびデータに誤りがないか確認します。
CytExpert nano:VSSC1チャネルでのEVサンプルと標準粒子のデータ取得
a. QC合格
b. Sensitivity Monitor合格
c. VSSC2が測定したハーフアングルのFit Confidenceが>90%
d. ベースラインモニタリングでCytoFLEX nanoが清浄であることを確認
e. タイトレーションにてサンプルの希釈率を決定し、凝集していないことを確認
Note: 80 nmおよび 125 nmビーズの散乱強度のMedianと上述の方法で決定したハーフアングルによって、80 nmおよび125 nmの標準ビーズと実験サンプルの両方を測定した時のゲインでのキャリブレーション曲線が設定されます。ご自身のアプリケーションで決定したゲインを使用することは可能ですが、少なくともQC Gain設定も同時に使用することを推奨します。
FCMPASS:VSSC2から近似したハーフアングルを用いたVSSC1データのキャリブレーション
a. データ取得日
b. データセット名(キャリブレーションに既知のハーフアングルを使用したことが分かるような名前を付ける)
c. データセットのメモ
a. EV直径– High RI, 1.4060
b. EV直径 – Average RI, 1.3859
c. EV直径 – Low RI, 1.3658
d. EV直径 – rEV CS RI, 1.42
e. ユーザーがサンプルから得たRI用として、Core-shellチャネルをナノメートル単位で作成し追加することができます。「+」をクリックして、文献、あるいは実証から得たカスタムRIを追加します。
f. 固体球粒子のRIが既知の場合、Solid-sphereチャネルをナノメートル単位で作成し追加することができます。「+」をクリックして、文献、あるいは実証から得たカスタムRIを追加します。
Note: カスタムRIを追加する場合は、そのカスタムRIに固有のパラメータ名を入力します。
Note: EVの不均一な分散をモデル化するため、FCMPASSは、シェル厚が5 nmのコアシェルモデルと、低~高の3つのRI値(RI=1.3658~ RI=1.4060)を用いたEV専用のRIキャリブレーションパラメータをデフォルト表示します。「rEV」はSigma-Aldrich社製のExosome標準蛍光粒子(製品番号:SAE0913)です。
Note: 問題が認められる場合は、Medianデータの入力ミスがないか確認します。Mie散乱領域の標準粒子サイズの値が入力されていないと、キャリブレーションに影響する可能性があります。データの入力やチャネルキャリブレーションの選択が正確でない場合も、失敗の原因となることがあります。
CytExpert nano:キャリブレーション済みCytoFLEX nano.FCSファイルのインポート
おわりに
CytoFLEX nanoで取得したデータの標準単位化は、ナノ粒子研究データの価値と意義を高めます。標準単位化によって、装置間だけでなく実験室間での比較も可能になります。CytoFLEX nanoとFCMPASSのようなキャリブレーションソフトウエアを利用することは、EVの科学的研究における再現性、検証可能性、トレーサビリティを確保するためにMISEVおよびMIFlowCyt-EVが提供する指針の一環でもあります。
FCMPASSの使用方法の詳細は、https://docs.fcmpass.com/からメーカー提供のFCMPASS関連文書をご参照ください。
参考文献
- Welsh, Joshua A, et al. “MIFlowCyt-EV: A Framework for Standardized Reporting of Extracellular Vesicle Flow Cytometry Experiments.” J Extracell Vesicles, vol. 9, no. 1, 2020, https://doi.org/10.1080/2 0013078.2020.1713526.
- Welsh, Joshua A, et al. “FCMPASS Software Aids Extracellular Vesicle Light Scatter Standardization.” Cytometry A, vol. 97, no. 6, 2020, pp. 569-581, https://doi.org/10.1002/cyto.a.23782.
- FCMPASS Resources: Known Angle Scatter Calibration https://resources.fcmpass.com/examples/knownangle- scatter-calibration
- CytoFLEX nano Instructions for Use (Software Version 1.0) https://www.beckman.com/techdocs/ C89615AA/wsr-422373
- A New Approach to Nanoscale Flow Cytometry with the CytoFLEX nano Analyzer https://www. beckman.com/resources/reading-material/application-notes/cytoflex-nano-flow-cytometer-the-newapproach- to-nanoscale-flow-cytometry
- Welsh JA, Horak P, Wilkinson JS, Ford VJ, Jones JC, Smith D, Holloway JA, Englyst NA., FCM(PASS) Software Aids Extracellular Vesicle Light Scatter Standardization, Cytometry A, 2019, DOI: 10.1002/ cyto.a.23782
- Welsh JA, Goberdhan DCI, O’Driscoll L, Buzas EI, Blenkiron C, Bussolati B, Cai H, Di Vizio D, Driedonks TAP, Erdbrügger U, Falcon-Perez JM, Fu QL, Hill AF, Lenassi M, Lim SK, Mahoney MG, Mohanty S, Möller A, Nieuwland R, Ochiya T, Sahoo S, Torrecilhas AC, Zheng L, Zijlstra A, Abuelreich S, Bagabas R, Bergese P, Bridges EM, Brucale M, Burger D, Carney RP, Cocucci E, Crescitelli R, Hanser E, Harris AL, Haughey NJ, Hendrix A, Ivanov AR, Jovanovic-Talisman T, Kruh-Garcia NA, Ku’ulei-Lyn Faustino V, Kyburz D, Lässer C, Lennon KM, Lötvall J, Maddox AL, Martens-Uzunova ES, Mizenko RR, Newman LA, Ridolfi A, Rohde E, Rojalin T, Rowland A, Saftics A, Sandau US, Saugstad JA, Shekari F, Swift S, Ter- Ovanesyan D, Tosar JP, Useckaite Z, Valle F, Varga Z, van der Pol E, van Herwijnen MJC, Wauben MHM, Wehman AM, Williams S, Zendrini A, Zimmerman AJ; MISEV Consortium; Théry C, Witwer KW. Minimal information for studies of extracellular vesicles (MISEV2023): From basic to advanced approaches. J Extracell Vesicles. 2024 Feb;13(2):e12404. doi: 10.1002/jev2.12404.
- Cook S, Tang VA, Lannigan J, Jones JC, Welsh JA. Quantitative flow cytometry enables end-toend optimization of cross-platform extracellular vesicle studies. Cell Rep Methods. 2023 Dec 18;3(12):100664. doi: 10.1016/j.crmeth.2023.100664
































